Factores importantes
como base para el mejoramiento genético en el cultivo de la piña (Anannas comosus var. comosus)
Andrés Zúñiga Orozco1,
Ayerin
Carrodeguas Gonzalez2
1.
Docente
e Investigador. Carrera Ingeniería Agronómica. Universidad Estatal a Distancia
(UNED). San José, Costa Rica, azunigao@uned.ac.cr
2.
Investigadora.
Instituto de Investigaciones Hortícolas Liliana Dimitrova. Mayabeque. Cuba, genetica2@lilliana.co.cu
Recibido: 18 de junio de 2020 Aceptado: 22 de octubre de 2020
RESUMEN
El cultivo de la piña (Anannas comosus L. var. comosus) es una fuente económica y de
empleo
importante en países tropicales y africanos. A pesar de los esfuerzos que se
han realizado por parte de centros de investigación el número de variedades que
se han producido e implementado en los últimos 100 años no ha generado gran
variabilidad de nuevos materiales genéticos; en la mayoría de los casos se han
restringido a evaluaciones de germoplasma, uso local y no han generado
variedades que se puedan adaptar a otras condiciones agroecológicas. En la
siguiente revisión se realiza una investigación de aspectos biológicos,
genéticos y moleculares por contemplar para llevar a cabo un proyecto de
mejoramiento genético en piña, con el fin de mejorar procesos y alcanzar los
objetivos de mejora en un tiempo más reducido.
Palabras
clave:
fitomejoramiento, piña, selección, cruzamiento, cultivo.
ABSTRACT
The cultivation of pineapple (Anannas comosus L. var. comosus) is an important economic and
employment source in tropical and African countries. Despite the efforts made
by research centers, the number of varieties that have been produced and
implemented in the last 100 years has not generated great variability in new
genetic materials; in most cases they have been restricted to germplasm
evaluations, local use and have not generated varieties that can be adapted to
other agroecological conditions. In the following review, an investigation of
biological, genetic and molecular aspects is carried out to be considered in
order to carry out a genetic improvement project in pineapple, in order to
improve processes and achieve the improvement objectives in a shorter time.
Key words: plant
breeding, pineapple, selection, crossing, cultivation.
Introducción
Con cerca de 200 especies
conocidas, distribuidas en 50 géneros, la piña (Ananas comosus L.) pertenece
a la familia Bromeliaceae (Jiménez, 1996) es la segunda fruta tropical
más consumida en el mundo. De acuerdo con cifras de la FAO, para el año 2014 la
producción de este fruto representaría en el mundo el 23% de la fruta tropical
cosechada (Superintendencia Industria y Comercio, 2015). La piña es originaria
de América del Sur, del centro y sureste de Brasil, así como del noreste de
Argentina y Paraguay. Ha sido seleccionada, desarrollada y domesticada durante
muchos siglos de cultivo. En la actualidad los frutos de piña y sus derivados
tienen gran importancia económica en las regiones tropicales y subtropicales
del mundo. Además de su popular consumo como fruta fresca, también se utiliza
para la producción de etanol, bromelina, fibras, fármacos y para la
alimentación animal (Nuñez 2011, Crestani et
al., 2010 y Santana et al.,
2013).
Los métodos de mejoramiento
genético de los cultivos parecen haber adquirido recientemente un carácter de
novedad, sin embrago, los registros históricos demuestran que el mejoramiento
se practicó desde que el hombre comenzó a seleccionar las mejores plantas en
los cultivos. Actualmente, el mejoramiento genético de las plantas se basa en
una completa comprensión y aplicación de los principios de la genética., así
como también exige el conocimiento de las enfermedades de las plantas y su
epidemiología y los factores que afectan su adaptación (Borém, Condori y
Miranda 2008). Adicionalmente, requiere mucha constancia y observación para
lograr un correcto análisis los datos.
Teniendo en cuenta la importancia de
implementar una estrategia para el mejoramiento genético del cultivo en
cuestión, se realizará una revisión sobre las bases genéticas y factores
involucrados en la especie Ananas comosus
var. comosus y se discutirán posibles estrategias que permitan el desarrollo de
nuevos cultivares.
Germoplasma
de piña,
un recurso invaluable con matices de
erosión genética: actualmente, la poca diversidad genética de la piña ha
afectado su producción y se ha convertido en un desincentivo para los entes de
financiamiento. La erosión genética del género Ananas se debe a la sustitución de cultivares nativos por
cultivares mejorados y a la acelerada tala de vegetación que ocurre en las
regiones consideradas como centros de diversidad. La utilización de pocos
cultivares es considerado un problema a ser resuelto por medio de mejoramiento
genético (Cabral et al., 1997), pero
para desarrollar esta actividad, según Delgado y Arango (2015), es prioritario
ampliar su base genética. Esto se podría
conseguir mediante la evaluación de diferentes recursos genéticos nativos con
el fin de identificar caracteres potencialmente útiles para el mejoramiento
(Coppens d’Eeckenbrugge, 1996).
Se han realizado importantes
esfuerzos en investigación para la mejora genética de este cultivo, a pesar de
que muchos de ellos no han ofrecido resultados concretos. Por ejemplo, durante
el período 1935-1972, fue realizado un estudio por el Pineapple Research
Institute (PRI) de Hawái, en el cual crearon y evaluaron más de 100 000
híbridos de piña (Williams y Flesh, 1993 y Collins, 1960). A pesar de que
obtuvieron variedades resistentes a Phytophthora
sp., con altos grados brix, sabores de pulpa novedosos y muchos otros
caracteres de importancia agronómica, siempre fueron rechazados por algún
carácter no deseado en comparación con la variedad comercial Cayena, además se
hizo poco uso de la estabilización de líneas puras. Lo anterior lamentablemente
revela como la aproximación y abordaje de los programas de mejoramiento durante
el último siglo no han brindado los resultados esperados para esta especie.
Para comenzar un programa de
mejoramiento genético, un factor clave es la disposición de un banco de
germoplasma debidamente caracterizado, ya sea a nivel morfológico o molecular,
para evaluar su diversidad genética y así definir la estrategia a adoptar en un
programa de cría o mejoramiento (Delgado y Arango 1995; Vieira et al., 2013). Se han realizado estudios
de caracterización de germoplasma de piña en Cuba por Isidrón et al. (2003) y Colombia
(Delgado-Huertas y Arango Wiesner 2015), así como en Brasil por el EMBRAPA
(Empresa Brasileña de Investigación Agropecuaria) y en el CIRAD (Centre de
Coopération Internationale en Recherche Agronomique pour le Développement) de
Francia. Para la caracterización y evaluación de genotipos se genera un
conjunto de datos de diferentes categorías (cualitativas y cuantitativas), los
cuales deben manejarse adecuadamente, ya que de lo contrario puede ser un
factor que dificulte el análisis e interpretación de los resultados. Debido a
lo anterior, es de mucha importancia que los valores obtenidos sean organizados
dentro de un coeficiente que mida la distancia entre ellos (Vidor et al., 2015). En este sentido, el
algoritmo propuesto por el coeficiente de Gower es muy útil para identificar la
similitud entre las diferentes accesiones (Cruz, Ferreira y Pessoni, 2011). Los
diferentes genotipos se encuentran en los recursos fitogenéticos ya sea
institucionales o privados, debido a que, en condiciones naturales, como se
explicó anteriormente, han sido sumamente afectados. Algunos autores han
propuesto una metodología para evaluar germoplasma en piña (Delgado-Huertas y
Arango-Wiesner, 2015)
Sistema
Reproductivo “anti-incesto”: en la piña conviven dos tipos de reproducción bien
definidos, sexual (en este caso alógama) y asexual. Para producción comercial,
la propagación es meramente asexual, se usa la selección de corona del fruto,
los hijos y los brotes vegetativos que la misma planta produce. Con el fin de
depurar y conservar la pureza genética de las variedades utilizadas, es
necesario desechar todo el material propagativo procedente de plantas enfermas
y con malformaciones de origen genético como: corona múltiple; hojas muy
espinosas, cintura y cuello. La uniformidad genética, sanitaria y del peso del
material de propagación, es uno de los factores que sostiene la productividad
en las plantaciones de piña (Superintendencia Industria y Comercio, 2015 y
Jiménez, 1996).
Por otra parte, la reproducción
sexual en este cultivo se usa para la hibridación intravarietal, la cual genera
variabilidad genética debido a la heterocigosis y posibilidad de usar el vigor
híbrido de sus parentales, así como también permite desarrollar líneas puras en
uno o varios caracteres de interés económico (Delgado y Arango, 1995). El tipo
de reproducción sexual que ocurre en piña se conoce como alogamia, la cual
consiste en la polinización cruzada y fecundación entre individuos
genéticamente diferentes. Este tipo de reproducción favorece la generación
constante de variabilidad genética en las poblaciones. Existen mecanismos que
favorecen la polinización cruzada en plantas, tales como la
autoincompatibilidad, heterostilia, hercogamia y dioecia (Delgado y Arango,
1995; Coppens D’eeckenbruge y Duval, 1995). La contribución de mutaciones
somáticas puede parecer importante para algún carácter económico, pero es
despreciable en comparación con los efectos de recombinación resultantes de la
reproducción sexual, sin embargo, ésta última está limitada por la
autoincompatibilidad, por lo que el mejorador debe lidiar con este efecto y a
su vez, buscar estrategias para superar esta dificultad.
Sistema
de autoincompatibilidad: se
ha comprobado que la incompatibilidad en piña es del tipo gametofítico, es
decir, depende de la constitución genética del gametofito. En otras palabras,
ocurre una inhibición del tubo polínico después de la fecundación según el
alelo presente en el polen tal y como se muestra en la figura 1. Esto se debe a que el genotipo del grano de
polen coincide en un alelo con el genotipo del estilo (Coppens d’Eeckenbrugge et al. 1997, Cabral et al. 2000).
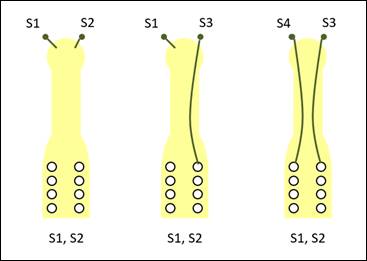
Figura 1. Sistema de
incompatibilidad gametofítica en piña (A.comosus
var. comosus). S= alelo. Fuente: Elaboración propia.
Este sistema promueve la
polinización cruzada, y provoca una mezcla muy heterogénea de genes en la
descendencia, la cual es aprovechada para adaptarse a diferentes condiciones
bióticas y abióticas. Por estas razones este mecanismo asegura más la sobrevivencia
de la especie que los mecanismos autógamos, ante posibles cambios en las
condiciones ambientales. Sin embrago, en la familia Bromeliaceae, se han
encontrado mutantes, como es el caso de Cayena
lisa, que son capaces de producir semillas por autofecundación. Otro
ejemplo es Pseudananas sagenarius una especie autofértil (Collins, 1960). Existe
una alta compatibilidad intraespecífica e interespecífica, lo que posibilita la
hibridación y el cruce entre diferentes cultivares. La semilla proveniente de
cruces, en la mayoría de casos es perfectamente viable, se reporta un periodo
de latencia de 6 meses, pero incluso en condiciones de conservación óptima se
ha registrado germinación hasta después de 2 años (Loison-Cabot, 1992).
Morfología
y citología floral:
para la reproducción sexual y mejoramiento genético es necesario conocer la
morfología de la flor, la cual se mantiene constante en el género Ananas, aunque se observan variaciones
en el tamaño de la inflorescencia y en las partes florales, como el número de
óvulos (Coppens d`Eckenbrugge y Duval 1995). Después de la polinización, la
flor sufre transformaciones para dar lugar al fruto. Como resultado de dichas
trasformaciones, la flor se convierte en un escudete octogonal de cubierta
dura, formada por la fusión del ápice de la bráctea y los tres sépalos, que
formará la dura piel cerúlea y espinosa del fruto (Polanco, 2017).
Polinizadores: se ha reportado como
polinizadores naturales algunos grupos de insectos como himenópteros, dípteros,
lepidópteros como Colias alfacariensis
y algunos coleópteros como Asclera
ruficolis. Además, aves como los colibríes, e incluso algunos mamíferos
como murciélagos y algunos pocos casos monos y ratones. Adicionalmente, se
pueden mencionar polinizadores naturales abióticos como el agua y el viento.
Ploidía:
A. comosus se ha reportado como
una especie diploide (2n) con 50 cromosomas (Collins y Kerns 1931, Marchant
1967, Collins 1960). Están reportados en la naturaleza triploides y
tetraploides, estos últimos como es esperable en otras especies vegetales, son
plantas con órganos más grandes y de crecimiento más lento que los 2n.
Recientemente también se reporta un tamaño estimado de 2,470 cM en longitud de
genoma (Carlier et al., 2012)
Fertilidad
reproductiva: En
general, la producción de semillas es baja, a excepción de algunos casos, donde
la habilidad combinatoria específica permite alguna combinación entre
parentales que produzca gran cantidad de semilla. En el estudio de Coppens
d`Eckenbrugge et al. (1993) se
determinó que la fertilidad fue baja en las variedades Cayena, Española Roja,
Queen, Singapore Spanish y Perola. En estos casos menos del 5% de los óvulos
producían semilla en condiciones de alopolinización libre, lo cual corresponde
a menos de 2 semillas por flor. Las variedades del grupo Mordiona (Perolera,
Rondon y Primavera) fueron medianamente fértiles con 2-5 semillas por flor, lo
que corresponde a 4-11% de fertilidad relativa. Las especies silvestres fueron
las más fértiles, con una fertilidad relativa para A. nanus de 6% y de 35-45% para A.
ananassoides, A. parguazensis y A. bracteatus. En A. comosus la baja fertilidad se ve compensada por una gran
cantidad de flores por inflorescencia por lo que hay oportunidad para hacer
mejoramiento convencional sin restricción por número de flores y cantidad de
semilla, pero se debe hacer una buena cantidad de polinizaciones (más de 30
polinizaciones por cruce). También se debe tener en cuenta que la mayor
cantidad de semillas se obtiene en los estratos bajos de la inflorescencia.
Incluso cuando un parental es muy valioso y se quiere emplear para hacer varios cruces, se puede usar la mitad
de la inflorescencia por los lados opuestos de una cara y de esta manera
aprovechar un fruto para dos parentales.
Genética
y variedades comerciales: La variedad más importante para exportación es MD-2,
debido a que posee un sabor y contextura agradable. Esta variedad fue promovida
en Costa Rica, pero fue desarrollada en Hawaii por el Pineapple Research
Institute (PRI). Existen otras variedades importantes en el mundo tales como la
Cayena Lisa conocida como hawaiana, la cual es apta para procesamiento y
consumo fresco. Cayena lisa es la segunda variedad de exportación, sin embargo,
es cada vez más desplazada por MD-2. A partir de la selección de Cayena Lisa
surgió Champaka F-153 en la India y fue evaluada en Hawaii. Se conocen otras
variedades que se cultivan en otras latitudes tales como: española roja,
Mauritus, PR-1-67, Cabezona, Pernambuco, Montufar, Abacaxi, Ripley, James
Queen, Queen, Spanish Jewel, Sugar Loaf, Singapore Spanish, Masmerah, Monte
Lirio (proveniente de Centroamérica), Perolera, Barón de Rothschild, Brecheche,
Burmanguesa, Maipure y Rondon (Muravi et
al., 2018; Isidrón, 2003, Isidrón et al., 2003, Castiñeira et al., 2000,
Mazani y Segovia, 2001, Duval et al., 1997)
Características
dominantes y recesivas de la piña: de acuerdo con Delgado y Arango
(2013), una de las características más notables que permite la diferenciación
de variedades en Ananas es la
distribución de espinas en hojas. Collins y Kerns (1946) determinaron que las
espinas son un carácter cualitativo gobernado por dos genes de herencia
sencilla, “s” y “p”. Para el caso de la Cayena donde el fenotipo es
semiespinoso y otros cultivares sin espinas, el alelo presente es “S”; mientras
que las variedades con el alelo “s” en su condición homocigota recesivo,
presentan espinas a lo largo de todo el borde. Del grupo de las especies menos
evolucionadas encontramos las que poseen más espinas, entre ellas la Ornamental
común, Roja y la Mayanez. Del grupo de distribución regular donde las espinas
se encuentran sólo atrás de la punta y cerca de la base (semiespinoso) se
encuentran la Clavo, MD-2 y Cayena Lisa, las cuales además fueron mejoradas
genéticamente por métodos convencionales. Por último, se encuentra el grupo
constituido por las especies que no tienen espinas en los márgenes como la
Perolera y Mitú. Según Delgado y Arango (2013), al analizar cuatro genotipos de
piña, se comprobó que el carácter forma del fruto redonda es dominante sobre
otros como la forma cilíndrica. Además,
se logró demostrar que la especie Mitú es capaz de donar genes para menor
cantidad de espinas, la Cayena Lisa tiene el potencial de heredar genotipos
aprovechables para el mejoramiento del tamaño del fruto, mientras la MD-2 junto
a la Mayanez fueron los mejores genotipos para aumentar grados Brix.
Adicionalmente, el híbrido MD-2 presentó un pedúnculo más corto con 21.1 cm,
una diferencia muy significativa con respecto a los otros tres genotipos. Se ha
encontrado también que la resistencia a Fusarium
subglutinans y el color rojo de las hojas son caracteres dominantes de
herencia cualitativa sencilla de un solo gen (Cabral et al., 1997). Dicho color de las hojas es dominante ante el color
verde y es muy probable que esto determine también el color del fruto. En el
caso de iniciar proyectos enfocados en piña de cáscara roja, se aclara que lo
anterior está en función de los parentales usados inicialmente, dado que,
dependiendo de las variedades usadas los resultados pueden o no cambiar.
Estrategia
de cruzamiento y selección: tradicionalmente, algunas de las estrategias de
mejoramiento que se han utilizado en piña se han basado en la selección clonal
y en métodos de recombinación sexual para la obtención de híbridos. En la
selección clonal se utilizan diferentes procesos, entre los que se destacan:
depuración de plantas defectuosas en campo, selección y multiplicación de
plantas élite y prueba de clones resultantes con selección final sobre varios
ciclos de cultivo (Superintendencia Industria y Comercio, 2015). A pesar de lo
anterior, la estrategia más efectiva es el uso de la reproducción sexual para
generar variabilidad y a partir de ahí comenzar con genotipos variables. No se
podría decir que exista una estrategia de cruzamiento ideal o una receta
establecida; usualmente en los cultivos, las estrategias se trazan en función
de los objetivos, la heredabilidad de los caracteres, los recursos, la especie
vegetal, los mecanismos de reproducción y el resultado de las progenies. Sin
embargo, hay ciertos criterios prácticos que se pueden seguir para tomar las
decisiones correctas al momento de abordar el cruzamiento y selección, como por
ejemplo el contar con una amplia colección de germoplasma, realizar evaluaciones
de cada accesión previo a utilizarlos como parentales, realizar cruzamientos en
la mayor cantidad de parentales posible para aprovechar oportunidad de
floración, organizar bloques de cruzamiento y contemplar todas las tecnologías
actuales disponibles y accesibles que nos colaboren para modificar la expresión
de los fenotipos en un menor tiempo.
Usualmente al iniciar un programa
de mejora, se necesita contar con un banco de germoplasma caracterizado para no
tener que comenzar a generarlo mediante cruzamientos, sin embargo, se puede
realizar, solo que toma mucho tiempo. Para generar ese banco de germoplasma se
pueden hacer fácilmente polinizaciones entre diferentes especies del mismo
género (interespecíficas) e incluso en algunos casos se han realizado entre
géneros (intergenéricos), éstas últimas no son tan exitosas si la ploidía es
muy diferente entre ellas. Sin embargo,
se han registrado casos en los cuales se han llegado a obtener unas pocas
semillas con caracteres bastante interesantes para el mercado de la piña
ornamental propagado clonalmente. García y Serrano (2005) reportan híbridos
interespecíficos entre A. comosus x A. bracteatus, que son el resultado de
primeros esfuerzos por mejorar caracteres, por otra parte, algunos de los
híbridos F1 que se han logrado obtener, se han logrado al cruzar Primavera x
Perola, Cayena x Perola, Cayena x Sao Bento y Champaka x MD-2. Benega et al. (1997) reportan híbridos F1
intraespecíficos de A. comosus entre
los cultivares Cayenna Lisa y Española Roja.
Posteriormente para el proceso de
fijar caracteres en generaciones segregantes, es necesario crear líneas
endogámicas o puras para poder cambiar la frecuencia alélica y expresar los
fenotipos deseados, es un proceso muy lento puesto que, el ciclo de producción
de ésta especie puede extenderse desde semilla a fruta por alrededor de 2.5-3
años. Un error muy comúnmente que se comete es creer que, al tener parentales
con los caracteres deseados éstos van a ser heredados en la progenie siguiente,
sin embargo, esto rara vez ocurre, ya que, lo usual es que se produzcan nuevas
segregaciones en cada carácter al estar en su condición heterocigota. Hay que
entender a los caracteres como un cubo de “Rubik”, una vez que se alcanza una
cara de un color es muy posible que las otras caras se desordenen. En el mundo
de la genética la idea anterior se manifiesta cuando otro carácter muestra otro
fenotipo no deseado.
La única forma de obtener todos los
caracteres deseados mediante cruces sería que estos estuviesen ligados con una
correlación altamente positiva, así cuando se mueve un carácter hacia un
fenotipo deseado agronómicamente el resto de caracteres también lo harían. En
genética de poblaciones esto se conoce como selección por arrastre o
“hitchhiking” por su nombre en inglés, sin embargo, en la realidad éste fenómeno rara vez ocurre en caracteres
de interés, especialmente en piña donde la mayoría de caracteres son
cuantitativos. Todo lo anterior sugiere que el proceso de creación de líneas
puras debe ser cuantificado y descrito, ya que, en ocasiones solamente se toman
notas de campo y se olvida analizar los datos para observar las tendencias.
Además, no se debe descartar la biotecnología para la obtención de líneas
puras.
Un factor a tomar en cuenta en este
cultivo es la presencia de la depresión endogámica. En otros cultivos no
relacionados con piña, se puede citar una disminución en la producción de
semilla y un efecto muy marcado en el vigor de las plantas (Redmond y Stout
2018, Bertan et al. 2009, Maeda et al. 2005, Cabral et al, 2000), caso
de Caladium x bicolor, Impatiens walleriana, Zea mays, incluso
en animales se observa la pérdida de la aptitud combinatoria. En plantas
alógamas como es el caso de la piña, el efecto de la endogamia generalmente es
mayor que en autógamas puesto que evolutivamente no están acostumbradas a
fertilizarse con su propio polen o polen de plantas muy emparentadas. Al ser
plantas alógamas presentan un alto grado de rechazo a la homocigosis lo cual
está reportado por Coppens D’eeckenbruge y Duval (1995), sin embargo, tiene una
pequeña oportunidad de tener éxito en conseguir semilla autopolinizada (Cabral et al., 2003, Collins, 1960). Ésta
semilla, se puede usar hasta la F2 de inbreeding, puesto que después, tanto el
vigor de las plantas como la cantidad de semilla se reducen drásticamente, lo
cual hace que se detenga el proyecto y comúnmente se conoce como callejón sin
salida o “dead end”. En este punto es más recomendable iniciar los cruces con
plantas hermanas o retrocruces, una vez seleccionadas las mejores en la F1
hasta conseguir líneas puras para los caracteres de interés. Es importante
recalcar que al utilizar esta estrategia algunos caracteres se pueden ver
afectados negativamente como se mencionó anteriormente, sin embargo, es más
manejable y brinda como máximo 2 generaciones de inbreeding dependiendo de cada
progenie resultante.
Por otro lado, una estrategia no
muy utilizada a pesar de que puede ser efectiva, es el uso de la polinización
masal, ésta estrategia tiene como ventaja que es fácil, barata, requiere poco
espacio y puede uniformar caracteres hacia homocigosis rápidamente, también
puede ser usada en cruces iniciales para obtener variabilidad. Tiene como
desventaja que los caracteres con baja heredabilidad son difíciles de fijar,
también al no ser tan específico, produce que se fijen alelos tanto deseables
como indeseables en cada carácter y además no se le puede dar trazabilidad al
parental que genera buenos fenotipos. Pese a las desventajas ya mencionadas
podría ser una buena estrategia de polinización en caso de que se quiera fijar
un carácter rápidamente, por ejemplo, un color de cáscara, un pedúnculo corto,
un pedúnculo grueso, un mejor porcentaje de germinación para las progenies, un
contenido de brix alto, entre otros. Puede usarse especialmente en caracteres
de herencia sencilla como los cualitativos mencionados en la sección de
caracteres dominantes y recesivos, y, además, siempre y cuando la heredabilidad
del carácter sea alta.
Finalmente, los retrocruces han
sido muy utilizados en piña con la creencia de que el carácter o caracteres que
se quieren ingresar en la variedad comercial se van a ver manifestados en la F1,
incluso se comete el error de seguir retrocruzando hasta generaciones
posteriores sin éxito. Lo anterior se debe a que no se hace un estudio para
averiguar la herencia del caracter objetivo previamente, así como su posterior
homocigosis, dando como resultado las acostumbradas segregaciones
“espontáneas”, sin embargo, este efecto es causado por el investigador. Así
pues, el retrocruce es una herramienta muy efectiva, pero se aclara que no debe
ser utilizada sin conocer la genética de los caracteres en estudio y el grado
de homocigosis. Para solventar esta situación se recomienda hacer algunos “test
crosses” o cruces de prueba entre líneas puras para observar la expresión
fenotípica y la herencia.
Marcadores
moleculares como herramientas para mejora del proceso de mejoramiento: en el campo de la
biotecnología, cada día se abren nuevas posibilidades, iniciando con la
caracterización de germoplasma a nivel del genoma, el cual fue secuenciado por
Myng et al. (2015) en las variedades F153 y MD-2, encontrando 27024 genes. También para la caracterización de germoplasma de piña y
observación de variabilidad de caracteres, se han utilizado marcadores
morfológicos, bioquímicos y moleculares. Con marcadores ISSR, Aradhya et al. (1994) realizaron un estudio en
un banco de germoplasma de Hawaii e identificaron 3 clusters mayoritarios en 15
accesiones basados en su variabilidad. Los marcadores moleculares, ya sean
dominantes o codominantes, son los más recomendados por el alto grado de
polimorfismo que permiten detectar, además permiten identificar genes
relacionados a caracteres de interés biológico y agronómico (Hayden et al. 2010). Usando marcadores AFLP,
Kato et al. (2005) caracterizaron 148
accesiones de A. comosus mantenidas
en una colección de piña en Hawai. Estos autores reportaron que no había
congruencia entre el fenotipo y la clasificación basada en este tipo de
marcadores moleculares por lo que no son los más adecuados para utilizar en
este cultivo.
Tapia
et al. (2005) caracterizaron
diferentes accesiones de germoplasma de piña utilizando iniciadores ISSR
(específicos), RAPD (no específicos) y microsatélites. Carlier et al. (2012) obtuvieron evidencias de
polimorfismo en los materiales haciendo un mapeo del genoma de la piña, además
con diferencias apreciables con respecto a las descripciones morfológicas, lo
cual hace que la herramienta de marcadores moleculares se vuelva indispensable
en cuanto a estudios de caracterización y filogenia en el género Ananas. Algunos genes importantes se
pueden identificar y comparar los polimorfismos a través de SNPs (single
nucleotide polimorfism). Los SNPs son la clase más
abundante de polimorfismos en el genoma de las plantas. La naturaleza dialélica
de los SNP asegura una tasa de error mucho más baja y facilita la comparación entre
laboratorios.
Actualmente la tecnología de
marcadores moleculares llamados microsatélites es una de las más utilizadas por
su nivel de especificidad, uso de codominancia y carácter multialélico. La
única desventaja que presenta este tipo de marcadores es que son difícilmente
automatizables, sin embargo, son bastante versátiles y aplicables en múltiples
estudios. Feng et al. (2013) hizo un
estimado de la diversidad de germoplasma en piña usando microsatélites. En este
estudio se crearon 4 grandes grupos de accesiones teniendo en cuenta las
similitudes moleculares, y se reportó que los grupos 1 y 2 fueron los más
cercanos. Este tipo de trabajos han creado una base sólida muy necesaria para
separar materiales genéticos y realizar cruzamientos. Los microsatélites
incluso pueden utilizarse para encontrar regiones de ligamiento de QTL
(Quantitative Trait Loci) en las cuales se pueda realizar un mapeo de regiones
del genoma involucradas en caracteres cuantitativos y detectar la presencia de
caracteres anticipadamente o selección temprana, de esta manera descartar
material con poco valor económico y haciendo más eficiente el proceso de
cruzamiento y selección.
Debido a estas
ventajas, en los últimos años, ha aumentado el uso de estos marcadores para la
identificación precisa de genotipos y el análisis de diversidad en cultivos
perennes (Zhou et al. 2015), como en
cacao (Ji et al. 2013), vid (Cabezas et al. 2011) y fresa (Ge et al. 2013). Al igual que otros
cultivos de horticultura perennes los estudios relacionados a la mejora
genética de la piña demandan el uso de este tipo de marcadores moleculares.
Estas aplicaciones incluyen, entre otras, la identificación de accesiones mal
etiquetadas, análisis de parentesco y de hermandad para el control de calidad
en programas de mejoramiento y semillas, y la producción de clones de alto
valor para el mercado (Zhou et al.
2015) y una aplicación altamente útil para el fitomejorador: la selección
temprana asistida por marcadores moleculares.
Bases
moleculares de la floración: existe actualmente con las nuevas tecnologías de
secuenciación bases de datos del genoma de la piña, así como de genes transcritos
y proteínas involucradas. Estudios previos han identificado genes expresándose
en la floración de A.comosus, tales
como FLOWERING LOCUS T (FT), LEAFY (LFY), PISTILLATA (PI), FT-LIKE y
AP1-LIKE (APETALA1), los cuales han mostrado regulación del meristemo y la
morfogénesis floral (Lv et al. 2012a, 2012b y 2016), sin embargo, es interesante citar el
trabajo de Wang et al. (2020) donde se realiza un análisis del transcriptoma,
de las estructuras florales y de la formación de la fruta, en éste estudio se
encontró una red molecular de genes interactuando para desarrollar el
crecimiento del órgano reproductivo (flor). Seis genes fueron identificados en
la expresión del desarrollo de óvulos y estambres, además el rol importante del
gen AcSBT1.8 en el desarrollo de los pétalos (Wang et al., 2020). Hay otras
investigaciones donde se correlacionan genes con el evento de floración, Chen
et al. (2019) encontraron a los genes PP2A y UBQ expresándose de manera estable
durante el evento reproductivo de floración, también se han encontrado otros
genes expresados de manera estable durante estrés abiótico, siendo PP2A y CYC
los más importantes. También otros genes como RAN, EF1α, PP2A relacionados
con la expresión metabólica debida a presencia de hormonas. Llama la atención
como el gen PP2A (protein phophatase 2 catalitic subunit 2 alfa, por su nombre
en inglés) está establemente expresado tanto para la floración, el estrés
abiótico y la síntesis de hormonas, convirtiéndose en un factor de
normalización, el cual también se ha identificado en otras especies (Zhu et
al., 2013 y Chen et al., 2014). El PP2A es un gen de la proteína fosfatasa
tipo 2A de la Serina/Treonina y está implicado en el transporte de auxinas en Arabidopsis (TAIR, 2020).
Más recientemente con la tecnología
CRISPR-Cas9 se abre otro abanico de posibilidades para caracteres de interés
que son difíciles de mejorar por fitomejoramiento tradicional. Además del
interés en la floración y su regulación hay otros caracteres tales como la
longitud del pedúnculo, por su importancia en el volcamiento de la fruta, y el
contenido de ácido ascórbico, por su utilidad para darle a la fruta una mayor
vida poscosecha.
Conclusiones
Después de describir algunos
tópicos de las bases genéticas de la piña, se describe en cada uno como podría
abordarse. Es importante mencionar que, independientemente del método a
utilizar, la clave de realizar un proceso exitoso es elegir el método más adecuado.
Para esto se deben de tener en cuenta: los recursos con los que cuente cada
institución, los resultados de las progenies que se vayan obteniendo y tener
claro que es un proyecto a mediano y largo plazo, en el cual, el seguimiento y
constancia son dos recursos importantes a tomar en cuenta. También es importante combinar técnicas
clásicas con biotecnológicas en función de buscar acortar procesos y por ende
el tiempo de obtención de resultados.
Cada vez es más frecuente tomar en
cuenta los trabajos realizados a nivel molecular para entender la expresión y
fisiología de la floración, la cual es crítica en función de inducir el
evento, controlar su expresión y mejorar el momento en el cual se quiere
obtener la flor, tanto con fines de mejoramiento como de producción, más
considerando los eventos de floración natural. Con este artículo de revisión
se define una base para emprender un proyecto de mejoramiento genético de
piña.
Agradecimientos
Nadia Fernández Picado de la Universidad
Estatal a Distancia, por su aporte para el desarrollo de este manuscrito.
Referencias
Acevedo-García, J.,
Spencer, D., Thieron, H., Reinstadler, A., Hammond-Kosack, K., Phillips, A.L. y
Panstruga, R. (2016). Mlo-based powdery mildew resistance in hexaploid bread
wheat generated by a non-transgenic tilling approach. Plant Biotechnology Journal, 15, 3, pp. 367-378.
Aradhya,
M.K., Zee, F. y Manshart, R.M. (1994). Isozyme variation in cultivated and wild
pineapple. Euphytica, 79,
87-99.
Basabe, L. y Angélica, G. (2010). La piña Ananas sativus schult (familia
Bromeliaceae). Recuperado de: http://fitomejoramientoenpina.blogspot.com/2010/09/
Bertan, I., Carvalho,
F.I.F. de., Oliveira,
A.C. de., Silva, J.A.G.
da., Benin, G., Hartwig, I., Schmidt,
D.A.M., Valerio, I.P., Fonseca, D.R.
da. y Silveira, G.
da. (2009).
Effects of heterosis and endogamy on agronomic important traits in wheat. Revista Ceres, 56, 6, pp. 753-763
Borém, A., Condori, M.
y Miranda, G. (2008). Mejoramiento de plantas. Viҫosa, MG, Brasil. Ed., Universidad Federal de
Viҫosa.
Cabral, J.R.S., Souza, A.S., De Matos, A.P. y Caldas, R.C. (2003). Efecto de la autofecundación en cultivares de piña. Diario de Fruticultura, Jaboticabal, 25, 1, pp. 184-185.
Cabral, J.R.S., de Matos, A.P. y Coppens d’Eeckenbrugge, G. (1997). Segregation for resistance to fusariose, leaf margin type and leaf colour from the EMBRAPA Pineapple Hybridization Programme. Acta Horticulturae, 425, pp. 193-200.
Cabral, J.R.S., Coppens d’Eeckenbrugge, G y de Matos,
A.P. (2000). Introduction of selfing in pineapple breeding. Acta Horticulturae. 529, pp. 165-168.
Cabrera, J., Alfonsin, R. y Galán, S. (2007). Introducción y evaluación preliminar del cultivar de piña tropical MD2 bajo invernadero en las Islas Canarias. Acta Hort., 48, pp. 693-696.
Carlier, J.D., Sousa, N.H., Santo, T.E., d’Eeckenbrugge, G.C. y Leitao, J.M. (2012). A genetic map of pineapple (Ananas comosus(L) Merr.) including SCAR, CAPS, SSR and EST-SSR markers. Molecular Breeding, 29, 1, pp. 245-260.
Cerrato, I. (2013). Panorama mundial de la piña.
Recuperado de: file:///C:/Users/dvalverdeb/Downloads/PANORAMA-MUNDIAL-DE-LA-PINA.pdf
Collins, J.L.
(1960). The pineapple, botany, utilization and cultivation. London, UK. Ed.,
Leonard Hill.
Collins, J.L. y
Kerns, K.R. (1931). Genetic studies of pineapple. I.A preliminary report upon the chromosome number and meiosis in
seven pineapple varieties (Ananas sativus L.) and in Bromelia
penguin. L. Journal of Heredity,
22, 139-142.
Collins, J.L. y
Kerns K.R. (1946). Inheritance of three leaf types in the pineapple. Journal
Heredity, 37, pp. 123-128.
Delgado-Huertas, H. y Arango-Wiesner, L.
(2015). Caracterización morfoagronómica de genotipos de piña (Ananas spp.) en
un suelo de terraza alta de Villavicencio. ORINOQUIA, 19, 2, pp. 153-165.
Marchant, C.J.
(1967). Chromosome evolution in the Bromeliacea. Kew Bulletin, 21, pp. 161-168.
Cabezas,
J.A., Ibanez, J., Lijavetzky, D., Vélez, D., Bravo, G., Rodriguez, V., Carreño,
I., Jermakow, A., Carreño, J., (2011). A 48 SNP set for grapevine cultivar
identification. BMC Plant Biol., 11,
153, DOI: https://doi.org/10.1186/1471-2229-11-153
Castiñeira, L., Fundora, Z., Shagarodsky, T., Fuentes, V., Barrios, O., Moreno, V., Sánchez, P., González, A. V., Martínez-Fuentes, A., García, M. y Martínez, A (2000). La conservación in situ de la variabilidad de plantas de cultivo en dos localidades de Cuba. Rev. Jardín Botánico, 21, 1, pp. 25-45.
Coppens d`Eckenbrugge, G.,
Duval, M.F. y Van Miegroet, M.F. (1993). Fertility and selfincompatibility in the genus
Ananas. Acta Horticulturae, 334,
45-51.
Coppens
d’Eeckenbrugge, G., Leal, F. y Duval, M.F. (1997). Germplasm resources of
pineapple. Horticultural Review, 21,
133-175.
Crestani, M., Barbieri, R.L. y Oliveira, A.C. (2010). De las Américas al Mundo - Origen, domesticación y dispersión del abacaxi. Ciencia Rural Santa María, 40, 6, 1473-1483.
Cruz, C.D., Ferreira, F.M., Pessoni, L.A. (2011). Biometria Aplicada al estudio de la diversidad genética, Vol. 1. Viçosa, Brasil. Editorial UFV.
Delgado, H. y Arango, L. (2015). Caracterización
morfoagronómica de genotipos de piña (Ananas
spp.) en un suelo de terraza alta de Villavicencio. ORINOQUIA, 19, 2, pp. 153-165.
Duval, M. F., Coppens, D’Eeckenbrugge, G., Ferreira,
F.R., Cabral, J.R.S. y Biancheti, L. de B. (1997). First results front joint EMBRAPA-CIRAD Ananas
germplasm collecting in Brazil and French Guyana. Acta Horticulturae, 425, pp. 137-144.
Nuñez, M. (2011). Estudio del caso del cultivo de piña (Ananas comosus). Recuperado de: http://fitomejoramientofca2011.blogspot.com/2011/04/estudio-de-caso-del-cultivo-de-pina.html
García, M.D. y Serrano, H. (2005). La piña, Ananas comosus (L.) Merr. (Bromeliaceae), algo más que un fruto dulce y jugoso. Universidad Autónoma Metropolitana. Revista Digital Contactos, N°56. Recuperado de: http://www.izt.uam.mx/newpage/contactos/anterior/n56ne/pina.pdf
García-Valencia L.E., Bravo-Alberto, C.E. y Cruz-García, F. (2013). Evitando el incesto en las plantas: control genético y bioquímico. TIP Revista Especializada en Ciencias Químico-Biológicas, 16, 1, pp. 57-65
Ge, A.J., Han, J. y Li, X.D.
(2013). Characterization
of SNPs in strawberry cultivars in China. Genet.
Mol. Res., 12, pp. 639-664.
Hayden, M.J., Tabone, T.L., Nguyen, T.M., Coventry, S., Keiper, F.J., Fox, R.L., Chalmers, K.J., Mather, D.E. y Eglinton, J.A. (2010). An informative set of SNP markers for molecular characterization of Australian barley germplasm. Crop Plant. Sci., 61, pp. 70-83.
Isidrón, M., Rosales, Y., Pifferrer, A., Cisneros, A., Benega, R. y Carvajal, C. (2003). Caracterización del germoplasma de piña colectado en Cuba mediante prospección nacional: localización, diversidad genética y situación actual. Cultivos Tropicales, 24, 1, pp. 65-71.
Isidrón, M. (2003). Germoplasma de piña: colecta, caracterización y conservación. Multiplicación de ecotipos de interés a la producción. Informe Final Proyecto. Programa Nacional Científico-Técnico de Mejoramiento Vegetal y Recursos Fitogenéticos No.: -01500057, Ciego de Ávila, Cuba.
Ji, K.,
Zhang, D., Motilal, L.A., Boccara, M., Lachenaud, P. y Meinhardt, L.W. (2013).
Genetic diversity and parentage in farmer varieties of cacao (Theobroma cacao L.) from Honduras and
Nicaragua as revealed by single nucleotide polymorphism (SNP) markers. Genet. Resour. Crop. Evol., 60, pp. 441-453.
Jiménez, D. (1996). El Cultivo de
la Piña de Exportación. Instituto del Trópico Humedo de Tabasco. México.
Recuperado de:
http://lapiniatropical.blogspot.com/2015/04/taxonomia-botanica-y-fisiologia-de-la.html
Kato,
C.Y., Nagai, C., Moore, P.H. (2005). Intra-specific DNA polymorphism in
pineapple (Ananas comosus (L.) Merr.)
assessed by AFLP markers. Genet. Resour.
Crop. Evol., 51, pp. 815-825.
Lv, L. L. et al.
(2012 a). Cloning and expression analysis of a PISTILLATA homologous gene from
pineapple (Ananas comosus L. Merr). Int J. Mol. Sci. 13, pp. 1039-1053. DOI:
https://doi.org/10.3390/ijms13011039
Lv, L. et al. (2012
b). Isolation and characterization of a FLOWERING LOCUS T homolog from
pineapple (Ananas comosus(L.) Merr). Gene, 505, pp. 368–373. DOI: 10.1016/j.gene.2012.06.011
Liu, C. H. y Fan, C. (2016). De novo transcriptome assembly of floral buds
of pineapple and identification of differentially expressed genes in response
to ethephon induction. Front. Plant Sci.,
7, 203. DOI: https://doi.org/10.3389/fpls.2016.00203
Loison-Cabot, C.
(1992). Origin, phylogeny and evolution of pineapples species. Fruits. 47, 1, pp. 25-32.
Maeda, J.M., Abreu, H.dos.S. y Silva, S.P. (2005). Occurrence of albinism and estimative of endogamy coefficient in a population of Euterpe edulis Mart. Floresta e Ambiente, 12, 2, pp. 71-74.
Maravi, J., Buendia, O., Alvarado, L., Borjas, R.,
Castro-Cepero, V. y Julca, A. (2018). Characterization of pineapple farms (Ananas comosus var. comosus) in Cuyani
Microbasin, Pichanaki District, Chanchamayo Province (Junín, Perú). Peruvian Journal of Agronomy, 2, 1, pp.
20- 27.
Mazani, E. y
Segovia, V. (2001). Colecta de germoplasma en la ecoregión
de la península de Paria, Estado de Sucre, Venezuela. Plant Genetic Newsletter, 126, pp. 17-20.
Ming, Ray; Van Buren, Robert; Wai, Ching Man; Tang, Haibao; Schatz, Michael C., et al…. (2015). The pineapple genome and the evolution of CAM photosynthesis. The pineapple genome and the evolution of CAM photosynthesis. Nat. Genet. 47, pp. 1435-1442.
Polanco, D.A. (2017). Piña, características de la planta, cultivo, propiedades y beneficios. Recuperado de: https://naturaleza.paradais-sphynx.com/plantas/tipos-de-frutas/pina-propiedades-ananas-comosus.htm
Redmond, C.M. y
Stout, J.C. (2018). Breeding system and
pollination ecology of a potentially invasive alien Clematis vitalba L. in Ireland. Journal of Plant Ecology, 11, 1, pp. 56-63.
Santana, L.L de A., Reinhardt, D.H., Cunha, G.P. y Caldas, R.C. (2001). Altas densidades de siembra en el cultivo de la piña cv. Smooth Cayenne, bajo condiciones de secano. Diario de plantas frutales Jaboticabal, 23, pp. 353-358.
Superintendencia de Industria y Comercio (2015). Piña:
Mejoramiento genético y propagación. Recuperado de: https://issuu.com/quioscosic/docs/pina_final
TAIR (2020). The Arabidopsis
information resource. Recuperado de: https://www.arabidopsis.org/servlets/TairObject?type=locus&name=At1g69960
Tapia, E., Guillén, H. y Gutierrez, M.A. (2005). Caracterización genética de materiales de piña (Ananas spp.) mediante RAPD e ISSR. Revista Fitotecnia Mexicana, 28, 3, pp. 187-194.
Tsuji, S.S. (2012). Análisis filogenético y patogénica del agente causal de la marchitez por Fusarium de la piña en Brasil. (Tesis de Maestría), Universidad Federal Rural de Pernambuco, Recife, Brasil.
Vieira, E.A., Fialho, J.F., Fonseca, K.G. y Carvalho, L.J. (2013). Caracterización fenotípica y molecular de accesos de mandioca de industria con potencial de adaptación a las condiciones del Cerrado de Brasil Central. Ciencias Agrícolas Londrina, 34, 2, pp. 567-582.
Vidor, A., Gonzaga, M., Krause, W., Azeredo, L.S. y Wandreilla, G. (2015). Caracterización Agronómica y divergencia genética entre accesos de Abacaxi, en las condiciones del estado de Mato Grosso, Brasil. Revista Brasileira de Fruticultura, 37, 4, pp. 952-960.
Wang, L., Li, Y., Jin, X., Liu,
L., Dai, X. et al… (2020).
Floral transcriptomes reveal gene networks in pineapple floral growth and fruit
development. Communications Biology, 3, 500, DOI: https://doi.org/10.1038/s42003-020-01235-2
Williams, D.D.F. y
Fleish, H. (1993). Historical review of pineapple breeding in Hawaii. Acta Horticulturae, 334, pp. 67-66.
Zhou,
L., Matsumoto, T., Tan, H.W., Meinhardt, L., Mischke, S., Wang, B. y Zhang,
D. (2015). Developing single nucleotide
polymorphism markers for the identification of pineapple (Ananas comosus) germplasm. Horticulture
Research, 2, 15056, DOI: https://doi.org/10.1038/hortres.2015.56
Zhu, J., Zhang, L., Li, W., Han, S.,
Yang, W. y Qi, L. (2013). Reference gene selection for quantitative real-time
PCR normalization in Caragana intermedia under different abiotic stress
conditions. PLoS One, 8, 1, e53196
DOI: https://doi.org/10.1371/journal.pone.0053196