Caracterización de bacterias ácido-lácticas aisladas de bioinsumos tipo bioles con capacidad productora de compuestos indólicos
Karla
Montero-Castro1,2![]() , Jéssica Montero-Zamora1
, Jéssica Montero-Zamora1![]() Melissa
Chaves Phillips1
Melissa
Chaves Phillips1![]() , Cristofer Orozco-Ortiz1
, Cristofer Orozco-Ortiz1![]() & Emanuel Araya-Valverde1
& Emanuel Araya-Valverde1![]()
1. Centro Nacional de Alta Tecnología, Laboratorio CENIBiot, San José 1174-1200 Calle, Costa Rica; kmregencia@outlook.com, je_1891@hotmail.com, mchaves125@gmail.com, cris0493@gmail.com, earaya@cenat.ac.cr
2. Instituto Tecnológico de Costa Rica, Maestría en Ciencia y Tecnología para la Sostenibilidad, 223-21001, Alajuela, San Carlos, Costa Rica.
Recibido 28-I-2024 ● Corregido 26-IV-2024 ● Aceptado 25-V-2024
DOI: https://doi.org/10.22458/urj.v16i1.5160
|
ABSTRACT. “Characterization of lactic acid bacteria isolated from two samples of biole-type bio-inputs with the production capacity of indolic compounds”. Introduction: Modern agriculture seeks to improve sustainability and reduce environmental impact, while meeting the nutritional needs of current and future generations. A promising strategy is the use of microbial biostimulants to increase crop yields and decrease dependence on agrochemicals and fertilizers. Among these biostimulants, those that use beneficial microorganisms such as plant growth-promoting bacteria stand out. We focused this study on isolating and identifying lactic acid bacteria present in artisanal bioles, and characterizing them by their ability to produce indolic compounds. The findings suggest that some lactic acid bacteria present in the two bioles samples can act as producers of indole compounds, such as 3-indoleacetic acid, which makes them a promising alternative for more sustainable and environmentally friendly agricultural practices. Objective: Isolate and identify lactic acid bacteria present in two samples of artisanal biole, and characterize them by their growth and their ability to produce indolic compounds. Methods: We use molecular techniques such as 16S rRNA gene analysis and multilocus typing sequencing analysis to identify strains of lactic acid bacteria present in artisanal biole. Subsequently, we characterized the isolated strains using growth kinetics and colorimetric tests for the detection of indolic compounds, and the quantification of said compounds using high-efficiency liquid chromatography (HPLC). Results: We identified three different genera of lactic acid bacteria species which were Lacticaseibacillus paracasei, Lactiplantibacillus sp., and Lacticaseibacillus pantheris. Lactiplantibacillus sp. stood out for their ability to produce 3-indoleacetic acid. (M2) and Lacticaseibacillus paracasei (M4) with a concentration of 16,75 µg/mL and 4,57 µg/mL, respectively. Conclusion: Our results suggest that some lactic acid bacteria present in artisanal bioles can act as producers of indolic compounds such as 3-indoleacetic acid, which makes them a promising alternative for more sustainable and environmentally friendly agricultural practices.
Keywords: microbiota, biotechnology, sustainability, crops, farmers. |
|
RESUMEN. Introducción: La agricultura moderna busca mejorar la sostenibilidad y reducir el impacto ambiental, al mismo tiempo que busca satisfacer las necesidades alimenticias de la las generaciones actuales y futuras. Una estrategia prometedora es el uso de bioestimulantes microbianos para incrementar los rendimientos de los cultivos y disminuir la dependencia de agroquímicos y fertilizantes. Dentro de estos bioestimulantes sobresalen los que utilizan microorganismos benéficos como las bacterias promotoras del crecimiento en plantas. Este estudio lo enfocamos en aislar e identificar bacterias ácido-lácticas presentes en bioles de fabricación artesanal, y caracterizarlas por su capacidad para producir compuestos indólicos. Los hallazgos sugieren que algunas bacterias ácido-lácticas presentes en las dos muestras de bioles pueden actuar como productoras de compuestos indólicos, como el ácido 3-indolacético, lo que las convierte en una alternativa prometedora para prácticas agrícolas más sostenibles y respetuosas con el ambiente. Objetivo: Aislar, e identificar bacterias ácido-lácticas presentes en dos muestras de bioles de fabricación artesanal, y caracterizarlas por su crecimiento y su capacidad para producir compuestos indólicos. Métodos: Utilizamos técnicas moleculares como el análisis del gen 16S rRNA y un análisis de secuenciación de tipificación multilocus para identificar las cepas de bacterias ácido-lácticas presentes en bioles de fabricación artesanal. Posteriormente, caracterizamos las cepas aisladas mediante cinéticas de crecimiento y pruebas colorimétricas para la detección de compuestos indólicos, y la cuantificación de dichos compuestos mediante cromatografía líquida de alta eficiencia (HPLC). Resultados: Identificamos tres géneros distintos de especies de bacterias ácido-lácticas los cuales fueron Lacticaseibacillus paracasei, Lactiplantibacillus sp., y Lacticaseibacillus pantheris. Destacaron por su capacidad para producir ácido 3-indolacético las especies Lactiplantibacillus sp. (M2) y Lacticaseibacillus paracasei (M4) con una concentración de 16,75µg/mL y 4,57µg/mL, respectivamente. Conclusión: Nuestros resultados sugieren que algunas de las bacterias ácido-lácticas presentes en los bioles de fabricación artesanal pueden actuar como productoras de compuesto indólicos como ácido 3-indolacético e indol, lo que las convierte en una alternativa prometedora para prácticas agrícolas más sostenibles y respetuosas con el ambiente.
Palabras clave: microbiota, biotecnología, sustentabilidad, cultivos, agricultores. |
Una estrategia prometedora para
lograrlo es el uso de insumos biológicos como los bioestimulantes a base de
materiales naturales con el fin de incrementar el crecimiento y productividad
de las plantas, al tiempo que reduce la dependencia de los agroquímicos y
fertilizantes sintéticos (Dong et al., 2020). ![]() Los
bioestimulantes vegetales se definen como “cualquier sustancia o microorganismo
que se aplica a las plantas con el objetivo de mejorar la eficiencia
nutricional, la tolerancia al estrés abiótico y/o las características de
calidad del cultivo, independientemente de su contenido de nutrientes” (du
Jardin, 2015). Se encuentran disponibles en gran variedad de formulaciones y
combinaciones, pero se clasifican según su fuente y contenido en sustancias
húmicas y fúlvicas, hidrolizados de proteínas y otros compuestos que contienen
nitrógeno, extractos de algas y botánicos, quitosano y otros biopolímeros,
compuestos inorgánicos, hongos beneficiosos y bacterias beneficiosas.
Los
bioestimulantes vegetales se definen como “cualquier sustancia o microorganismo
que se aplica a las plantas con el objetivo de mejorar la eficiencia
nutricional, la tolerancia al estrés abiótico y/o las características de
calidad del cultivo, independientemente de su contenido de nutrientes” (du
Jardin, 2015). Se encuentran disponibles en gran variedad de formulaciones y
combinaciones, pero se clasifican según su fuente y contenido en sustancias
húmicas y fúlvicas, hidrolizados de proteínas y otros compuestos que contienen
nitrógeno, extractos de algas y botánicos, quitosano y otros biopolímeros,
compuestos inorgánicos, hongos beneficiosos y bacterias beneficiosas.
Entre los bioestimulantes, los bioles se destacan como una opción popular y accesible para los agricultores. Los bioles son abonos líquidos orgánicos, producidos mediante fermentaciones anaeróbicas de materiales orgánicos como estiércol, plantas, microorganismos, melaza y suero de leche (como fuentes de energía y proteína, respectivamente), entre otros. Esta mezcla resulta en una solución rica en nutrientes y compuestos beneficiosos para las plantas (Restrepo, 2007; Solís-Oba et al., 2021), lo que le permite aportar a la planta o al suelo donde esté cultivada, nutrientes, materia orgánica, antibióticos, vitaminas, minerales, fitohormonas u otros componentes beneficiosos producto de su composición microbiológica, y de esta forma estimulan el crecimiento de las plantas y fomentan el metabolismo vegetal que protege a la planta de enfermedades y movilizan los nutrientes en las interacciones suelo-planta (Zagoya, 2013; Zanabria 2019). Su aplicación puede ser foliar, directa al suelo, a la raíz o la semilla, por aspersión o mediante el sistema de riego (Linares-Gabriel et al., 2017). Por ejemplo, en Costa Rica se han obtenido resultados prometedores al aplicar bioles artesanales en cultivos de lechuga y brócoli, donde reportaron un aumento significativo en la altura, peso y rendimiento de las plantas tratadas en comparación con el grupo de control (Xiu, 2018), así como otros estudios que han comprobado efectos positivos en los cultivos (Guerrero, 2022; Muñiz, 2023).
![]() Entre los microorganismos presentes en los bioles encontramos
las “Bacterias Promotoras del Crecimiento en Plantas” (BPCP), las
cuales desempeñan un papel crucial en la mejora de la productividad agrícola. Estos
géneros bacterianos, como Bacillus, Streptomyces, Pseudomonas y Lactobacillus,
poseen la capacidad de estimular el crecimiento de las plantas, mejorar la
absorción de nutrientes y protegerlas contra patógenos. (Grover et al., 2011;
Glick, 2012).
Entre los microorganismos presentes en los bioles encontramos
las “Bacterias Promotoras del Crecimiento en Plantas” (BPCP), las
cuales desempeñan un papel crucial en la mejora de la productividad agrícola. Estos
géneros bacterianos, como Bacillus, Streptomyces, Pseudomonas y Lactobacillus,
poseen la capacidad de estimular el crecimiento de las plantas, mejorar la
absorción de nutrientes y protegerlas contra patógenos. (Grover et al., 2011;
Glick, 2012).
Estas bacterias al interactuar con las plantas, puede inducir una serie de efectos beneficiosos que mejoran su crecimiento, rendimiento y resistencia a condiciones adversas. Además de promover la absorción de nutrientes esenciales, como el nitrógeno y el fósforo, algunas BPCP tienen la capacidad de producir fitohormonas, como las auxinas, que estimulan el desarrollo de raíces y brotes, y promueven la formación de flores y frutos (Glick, 2012). Asimismo, se ha observado que ciertas bacterias, como las del género Bacillus, pueden producir antibióticos y compuestos antimicrobianos que protegen a las plantas contra patógenos del suelo y enfermedades (Borris et al., 2018). Existen reportes de aplicación de BPCP, donde estimulan el crecimiento y el rendimiento en distintas especies de frutas, como la manzana, cereza dulce, cítricos, frambuesa, arándano, albaricoque y fresas, entre otras (Glick, 2012), mediante la incorporación al sistema suelo-planta de bacterias fijadoras de nitrógeno, bacterias solubilizadoras de fósforo, bacterias promotoras del crecimiento de las plantas mediante la producción de auxinas, y/o bacterias que suprimen patógenos de plantas (Mazid & Khan, 2014). No obstante, dentro de este conjunto de microorganismos beneficiosos, las bacterias ácido-lácticas (BAL) destacan por su potencial para promover el crecimiento de las plantas y mejorar su resistencia al estrés abiótico. Dada la presencia de suero de leche como componente en la elaboración de los bioles, se favorece la proliferación de BAL en este sistema. Las BAL, como miembros clave de la microbiota del suelo, han demostrado su capacidad para sintetizar compuestos beneficiosos, como el ácido indol acético, a partir de la biosíntesis del aminoácido triptófano (Trp), lo que contribuye significativamente al desarrollo saludable de las plantas (Compant et al., 2010).
Los beneficios de las BAL en la agricultura son conocidos por promover el crecimiento radicular de las plantas en presencia de Trp ya que libera una variedad de compuestos indólicos, lo que contribuyen de forma positiva en el mejoramiento de los cultivos, sus métodos de captación de nutrientes y la calidad del suelo (Compant et al., 2010; Naik et al., 2019). Kang et al. (2015), en su estudio muestra los efectos beneficiosos de la aplicación de Lactobacillus plantarum en cultivos de pepino, donde incrementó la longitud de los brotes, de la raíz y del peso seco y peso fresco. Por su parte, Panetto et al., (2023), reporta que la especie Lactobacillus acidophilus en cultivos de papa inducidos con Trp, muestra la capacidad de sintetizar sideróforos, y de esta forma promover el crecimiento de la planta produciendo concentraciones de AIA de 7,25µg/mL, solubiliza fósforo en una concentración de 5,58µg/mL y fija nitrógeno en una concentración de 0,5µg/mL. Comparando estos datos con los obtenidos por Turaeva et al., (2022), quién reportó las concentraciones de dos auxinas producidas por el cultivo de Lactobacillus plantarum suplementado con L-Trp, produciendo una concentración máxima de AIA de 1,850mg/mL, esto nos ayuda a confirmar que las BAL en presencia del aminoácido Trp producen pequeñas concentraciones de compuestos indólicos tipo AIA.
Este estudio se adentra en un campo de investigación que promete contribuir a la búsqueda de soluciones agrícolas más eficientes y amigables con el ambiente, es por esto que el objetivo de esta investigación es el aislamiento e identificación molecular de BAL de dos muestras de bioles de fabricación artesanal, y su caracterización mediante cinéticas de crecimiento y pruebas químicas cualitativas para comprobar la presencia de compuestos indólicos, así como el uso de métodos cromatográficos para la cuantificación de la producción de dichos compuestos.
MATERIALES Y MÉTODOS
Obtención y preparación de la muestra: Las cepas las aislamos de bioles de microorganismos de montaña (MM) elaborados en una finca agrícola de la zona de Zarcero 10,18561, -84,39076, Alajuela. Estas muestras las trasladamos en frío al Laboratorio CENIBiot-CeNAT donde las almacenamos a 4°C para su conservación y análisis.
Aislamiento de bacterias ácido lácticas: Basándonos en el protocolo modificado descrito por Mantilla (2007), tomamos un volumen de 10mL de cada muestra de biol, y realizamos diluciones seriadas 1/10 en agua destilada estéril hasta 10-7. Colocamos un volumen de 1mL de las concentraciones de 10-4 hasta 10-7, en placas de Petri con agar de Man, Rogosa & Sharpe (MRS) (Sigma-Aldrich, St. Louis, Missouri, USA) por esparcimiento con asa de Drikalski.
Las placas las incubamos a 37°C durante 48 horas en sistemas de jarras de anaerobiosis, donde colocamos sobres absorbentes de oxígeno para generar las condiciones de una atmósfera libre de oxígeno. Finalizado este periodo de crecimiento, seleccionamos las colonias con la morfología característica de una BAL. Las colonias seleccionadas las subcultivamos en tubos cónicos Falcon® con caldo MRS donde fueron incubados a 37°C durante 48 horas sin agitación, las cepas purificadas las almacenamos en glicerol al 20% (v/v) a -80°C y en liofilizados, con el fin de conservar la cepa original.
Secuenciación e identificación molecular de las bacterias aisladas presentes en las muestras de bioles con la región 16S rRNA: Realizamos una identificación preliminar por la reacción en cadena de la polimerasa (PCR) de la región 16S rRNA, utilizando dos primers universales, 27F (5’- AGA GTT TGA TCM TGG CTC AG-3’) y 1492R (5’-CGG TTA CCT TGT TAC GAC TT-3’), tal y como se describió en Rahmoune et al. (2017). La secuenciación de la región 16S rRNA, la realizamos utilizando el kit de secuenciación Big Dye Terminator V3.1 Cycle Sequencing Kit (ThermoFisher Scientific, USA), siguiendo las recomendaciones del fabricante. Los productos de la reacción de secuenciación los limpiamos con el kit BigDye X-Terminator, y los visualizamos mediante electroforesis capilar en el analizador genético SeqStudio Genetic Analyzer (Applied biosystems, Thermo Fisher Scientific, USA).
El análisis de las secuencias de la región 16S rRNA lo realizamos en los programas BioEdit (Hall, 1999), y FinchTV Geopiza, donde, se comparan las secuencias consenso obtenidas con secuencias genéticas utilizando la base de datos del National Center for Biotechnology Information (NCBI) (www.ncbi.com), utilizamos la herramienta Nucleotide Basic Local Alignment Search Tool (BlastN) (Altschul et al., 1990), para determinar las secuencias de mayor similitud (megablast).
Tipificación Multilocus de Secuencias (MLST): Seleccionamos cinco cepas representativas de cada género de las cepas aisladas mediante selección al azar, para un análisis de tipificación posterior por MLTS (Lacticaseibacillus paracasei, Lactiplantibacillus sp., Lacticaseibacillus pantheris y dos muestras identificadas previamente como Lacticaseibacillus paracasei), para esto se siguió la metodología descrita por WingChing et al., (2021), con modificaciones.
Amplificamos las regiones que corresponden a los genes: ribonucleótido trifosfato reductasa (nrdD, por sus siglas en inglés) (ubicación: 129 746…130 540), alfa-fosfoglucomutasa (pgm, por sus siglas en inglés) (ubicación:927 617…928 409), ADN polimerasa I (polA, por sus siglas en inglés) (ubicación:1 702 740…1 703 578), proteína reparadora del ADN (mutL, por sus siglas en inglés) (2 225 886…2 226 701), la subunidad alfa polimerasa (rpoA, por sus siglas en inglés) (2 458 334…2 459 129) y la metionil-tARN sintetasa (metRS, por sus siglas en inglés) (2 586 141…2 586 862). Seleccionamos fragmentos de 621 nt del gen metRS, 696 nt del gen mutL, 722 nucleótidos (nt) del gen nrdD, 691 nt del gen pgm y 728nt del gen polA. El perfil térmico lo desarrollamos con una desnaturalización inicial de 95°C por 5 minutos, luego 35 ciclos de 95°C por 30 segundos, el anillamiento a 56 y 60°C por 30 segundos, y una extensión a 72°C por 60s. La extensión final la realizamos a 72°C por 10 min. La limpieza de los productos de PCR la llevamos a cabo con el reactivo ExoSAP-IT (ExoSAP-IT PCR Product Cleanup, Applied Biosystem), siguiendo las indicaciones del fabricante. Realizamos la reacción de secuenciación utilizando el kit de secuenciación Big Dye Terminator V3.1 Cycle Sequencing Kit (ThermoFisher Scientific, USA) siguiendo el protocolo del fabricante. Los productos de la reacción de secuenciación lo limpiamos con el kit BigDye X-Terminator y los visualizamos en el analizador genético SeqStudio Genetic Analyzer (Applied biosystems, Thermo Fisher Scientific, USA).
Construimos una base de datos donde consideramos 23 aislamientos, los cuales incluimos en las cinco cepas estudiadas en esta investigación y 18 secuencias de aislamientos disponibles en el GenBank (www.ncbi.com), la herramienta ClustaIW Multiple Aligment la utilizamos con la finalidad de alinear las secuencias idénticas obtenidas por cada alelo y cada gen, con su respectiva secuencia de referencia, por lo que se hizo un alineamiento múltiple de secuencias entre las secuencias con alto porcentaje de similitud o referencia y las secuencias en estudio.
El árbol filogenético lo construimos con las cinco cepas estudiadas en esta investigación y 18 secuencias de aislamientos disponibles en el GenBank (www.ncbi.com), utilizando el programa MEGA11 (Molecular Evolutionary Genetics Analysis), versión 11, (Tamura et al., 2021), donde concatenamos todos los genes siguiendo el orden antes descrito. La filogenia la elaboramos utilizando el protocolo detallado por Vélez et al., (2015), con algunas modificaciones, utilizamos el modelo de sustitución de nucleótidos que mejor se ajustó, en este caso el de Kimura 2-parameter con distribución estadística de Nearest Neighbor (Abad, 2021).
Cinética de crecimiento de las cepas seleccionadas: Seleccionamos cinco cepas (identificadas como M1, M2, M3, M4 y M5, basado en los resultados de identificación molecular de la región 16S rRNA). Luego, preparamos una suspensión bacteriana cultivando cada una de las cepas seleccionadas en 10mL de caldo MRS, dejamos en incubación a 37°C durante 48h. La cinética de crecimiento la desarrollamos según lo indicado en Montero-Zamora et al. (2020); en una placa de fondo plano estéril de 96 pocillos, llenamos con 250µL por pocillo de muestra, utilizamos medio de cultivo MRS como control negativo y el Lactobacillus casei ATCC 393 como control positivo. La suspensión bacteriana la diluimos hasta un valor de absorbancia inicial de 0,05, medida a una longitud de onda de 600nm. La cinética de crecimiento la determinamos monitoreando la densidad óptica (DO600) a 37 °C durante 48 horas, con lecturas cada 5min en un lector de microplacas (Biotek, Winooski, VT, USA). Las cinéticas de crecimiento las desarrollamos en dos medios de cultivo, MRS sin suplementar, y MRS suplementado con 5mg/mL de L-Triptófano (L-Trp), con el fin de ver la relación de la presencia de este aminoácido en el comportamiento de las cepas estudiadas.
Diseño estadístico: El análisis estadístico de los datos lo desarrollamos según lo escrito en Montero-Zamora et al., (2020), en el cual describen que todos los análisis se realizaron por triplicado y los resultados se expresaron como media ± desviación estándar. Utilizamos el programa Minitab (www.minitab.com) para realizar un análisis de varianza unidireccional (ANOVA), y cuando sea significativo y/o se observen diferencias (P< 0,05), aplicamos las técnicas de Duncan´s Multiple Range Test (DMRT).
Tal como hemos mencionado anteriormente, las BAL con capacidad para producir ácido-3- indolacético (AIA) se ven favorecidas en presencia del aminoácido triptófano, es por esta razón que realizamos una nueva prueba de cinética de crecimiento, pero en este caso los cultivos bacterianos los disolvimos en el medio de cultivo caldo MRS suplementado con L-triptofano (L-triptofano, Sigma Aldrich) en una concentración de 5mg/mL. Todas las condiciones de las cinéticas anteriores, se mantienen, excepto el medio de cultivo sin suplementar.
Caracterización por método colorimétrico: La caracterización colorimétrica para determinar la presencia de compuestos indólicos se la realizamos a las cinco cepas M1, M2, M3, M4 y M5, al caldo MRS y a la cepa Lactobacillus casei ATCC 393 utilizada como control positivo. Este proceso lo realizamos siguiendo el protocolo modificado expuesto por Zúñiga, (2009), donde tomamos 1,5mL de muestra del caldo bacteriano, y lo centrifugamos a 16 278 x g por 5min. Cuidadosamente tomamos 1mL del sobrenadante, y lo transferimos a un nuevo tuvo cónico de 2mL donde lo mezclamos con 1mL del reactivo de Van Urk Salkowski utilizado para la identificación de compuestos indólicos (Ehman, 1977), lo incubamos a 30°C en oscuridad durante 30 min. Como control utilizamos 1mL de medio sin inocular y 1mL del reactivo de Salkowski, cumpliendo con el proceso anteriormente detallado. La presencia de compuestos indólicos la observamos por la aparición de colores rosado, rojo o naranja en las muestras analizadas (Mohite, 2013).
Cuantificación de auxinas mediante Cromatografía Líquida de Alta Resolución (HPLC, por sus siglas en inglés): En la preparación de la muestras para la cuantificación de compuestos indólicos de los aislamientos, utilizamos el método de filtración modificado reportado por Szkop y Bielawski, (2013), tomamos una alícuota de 1,5mL de caldo de cada cultivo bacteriano de cada una de las cepas y lo colocamos en un tubo Eppendorf® de 2mL, lo centrifugamos a 13 400rpm en una microcentrífuga (Eppendorf, Centrifuge 5424, Alemania) durante 10 minutos, posteriormente tomamos una alícuota de 500µL del sobrenadante, lo filtramos en columnas de C18 (LC-18 Supelguard™ Cartridge, Sigma Aldrich). Finalmente, el filtrado obtenido lo utilizamos en la cuantificación.
La cuantificación de compuestos indólicos producidos por las cepas la realizamos mediante cromatografía por HPLC en un cromatógrafo (Agilent 1200, USA) seguimos el protocolo descrito en Castillo et al. (2005), con algunas modificaciones. En este caso utilizamos una columna Luna 5U C18 (250 x 4,60mm Phenomenex). Como fase móvil utilizamos una mezcla de metanol y agua 1:1 (v/v), a una velocidad de flujo de 0,7mL/min y a temperatura ambiente. La detección la realizamos por fluorescencia usando una longitud de onda (λ) específica para cada patrón de compuesto indólico analizado, 220nm para el ácido 3-indolacético (AIA), 273nm para el indol y 226nm para el ácido 3-indol butírico (AIB), con el fin de conocer la posible ruta dependiente o independiente del Trp que siguen los aislamientos.
Las concentraciones de compuestos indólicos presentes en las cepas aisladas la determinamos a partir de la comparación con curvas estándar de los patrones de fitohormonas anteriormente descritos. A partir de cada solución madre de compuestos indólicos, preparamos curvas de calibración de ocho puntos de concentración de cada compuesto indólico, con valores de 0,001; 0,003; 0,005; 0,010; 0,030; 0,050; 0,100; 0,300µg/mL, en donde cada disolución la inyectamos por triplicado. Verificamos la existencia de una correlación lineal entre la relación de las áreas de los picos cromatográficos de cada compuesto indólico y las concentraciones correspondientes, para obtener la relación de mejor ajuste. La clasificación de las bacterias productoras en base a la concentración de AIA, se realizó según la propuesta de Khalid et al. (2004), como bajas, medianas y altas productoras. En el caso de las bajas productoras manejan concentraciones en un rango entre 1 – 10μg/mL, las medianas productoras maneja concentraciones de 11 – 20μg/mL, y las altas productoras concentraciones de 21 – 30μg/mL.
RESULTADOS
Identificamos un total de 23 cepas que en base a las secuencias del gen 16S rRNA y en comparación con los datos registrados en el GenBank (www.ncbi.com) confirmamos la presencia de los géneros Lacticaseibacillus, Lactiplantibacillus y Lactiobacillus. De estas cepas, nueve presentaron un 99,93% de similitud con Lacticaseibacillus paracasei y las denominamos grupo M1, seis secuencias las ubicamos en el grupo M2 y presentaron 100,00% y 99,80% de compatibilidad con Lactiplantibacillus plantarum y Lactiplantibacillus carotarum, respectivamente. En este caso solamente se reportó el género, ya que no se puede determinar la especie. Por su parte el grupo M3 estuvo representado por una sola cepa, la cual identificamos como Lacticaseibacillus pantheris con una similitud del 100,00%, en el último grupo seleccionamos siete cepas que se identificaron como Lacticaseibacillus paracasei con un 100,00% de similitud. Para las L. paracasei, se seleccionaron tres cepas (M1, M4 y M5), en el caso de M4 y M5 pertenecientes al último grupo, seleccionamos ambas dado que al comparar sus secuencias mostraron diferencias en aminoácidos en regiones específicas, pero ambas con un 100,00% de similitud. Por su parte, M1 obtuvimos un porcentaje de identidad de 99,93%, confirmando que las secuencias son diferentes, lo que nos generó el deseo de verificar si esos cambios representaban alguna diferencia funcional en las muestras (Tabla 1).
Determinación de la taxonomía utilizando el gen 16S rRNA de las especies de bacterias ácido-lácticas aisladas
|
Código |
NCBI |
Máxima identidad (%) |
Aislamientos similares |
|
M1 |
Lacticaseibacillus paracasei |
99,93 |
9 |
|
M2 |
Lactiplantibacillus sp. |
100,00 |
6 |
|
M3 |
Lacticaseibacillus pantheris |
100,00 |
1 |
|
M4 |
Lacticaseibacillus paracasei |
100,00 |
7 |
|
M5 |
Lacticaseibacillus paracasei |
100,00 |
|
|
M6 |
Lactobacillus casei ATCC 393 |
100,00 |
NA |
NA. No Aplica
Para identificación de las cepas en todas las pruebas posteriores, se utilizará la codificación mencionada anteriormente, donde M6 es el control positivo.
Realizamos el análisis de tipificación multilocus (MLST) para las cinco cepas seleccionadas, pero el árbol filogenético lo construimos a partir de las secuencias de los genes metRS, multL, nrdD, pgm y polA para tres de las cepas estudiadas, en este caso excluimos del análisis las cepas M2 y M3, ya que no contaban con secuencias de referencia para todos los fragmentos analizados. En base en lo anterior, el árbol filogenético lo construimos únicamente con las cepas M1, M4 y M5, e incluimos como referencia 18 secuencias de BAL de los géneros Lacticaseibacillus sp., Lactobacillus sp. con las especies L. casei y L. paracasei (Figura 1). Observamos que los aislamientos M4 y M5 identificados como Lacticaseibacillus paracasei en esta investigación, están ubicados en un mismo clado, junto a las referencias de Lacticaseibacillus paracasei. A su vez observamos que M1, la cual también lo identificamos como Lacticaseibacillus paracasei, la ubicamos dentro de otro clado diferente a las cepas anteriores, donde se agrupo junto con las referencias de Lactobacillus casei y Lactobacillus paracasei, lo que nos sugiere que para una clasificación correcta de esta cepa se requiere pruebas adicionales, como lo sería un análisis de genoma completo. Los otros clados que observamos, son agrupaciones de bacterias Lactobacillus casei y Lactobacillus paracasei, pero ninguno agrupo con las cepas estudiadas.
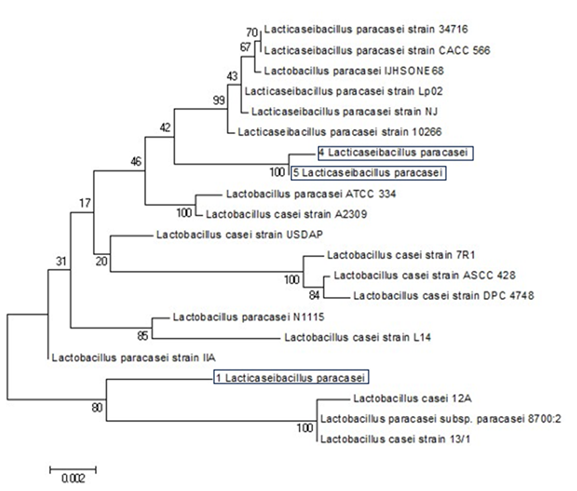
Fig. 1. Árbol filogenético de secuencias de bacterias ácido-lácticas aisladas de bioestimulantes costarricenses
Las cinéticas de crecimiento las realizamos comparando los sistemas con medio MRS y MRS suplementado con L-Trp para las cinco cepas aisladas y el control M6, obteniendo datos de DO600 máximo y tasa de crecimiento para las seis cepas (Tabla 2).
Observamos que M2 y M5 son estadísticamente significativas las que alcanzaron la mayor densidad óptica (DO600) en MRS sin L-Trp, con valores de 1,53 ± 0,03 y 1,40 ± 0,40, respectivamente, mientras que la cepa M4 mostró la mayor tasa de crecimiento en este medio, con un valor de 0,0047 ± 0,0003 (h-1), pero a pesar de esto, no produce la mayor biomasa (1,37 ± 0,05). Respecto a la M2, a pesar de presentar la mayor DO600, muestra la menor tasa de crecimiento de todas las muestras 0,0033 ± 0,0001 (h-1) estudiadas en este sistema.
Por otra parte, en presencia de L-Trp, observamos que las cepas M1 y M4 presentaron la mayor DO600 con valores de 1,45 ± 0,06 y 1,51 ± 0,06, respectivamente. Por su parte la cepa M5 mostró la menor densidad y tasa de crecimiento, reportando valores de 0,81± 0,04 y 0,10 ± 0,01, respectivamente
TABLA 2
Densidad óptica máxima, y tasa de crecimiento específica para la cinética de crecimiento de especies de BAL aislados de bioles microbianos en medio MRS y MRS suplementado con L-triptofano
|
Tratamiento |
MRS sin L-triptofano |
MRS suplementado con L-Triptofano |
||
|
Código |
Máximo OD600* |
Tasa crecimiento (h-1)* |
Máximo OD600* |
Tasa crecimiento (h-1)* |
|
M1 |
1,35 ± 0,06b, c |
0,0041 ± 0,0008b |
1,45 ± 0,06b |
0,25 ± 0,01a |
|
M2 |
1,53 ± 0,03a |
0,0033 ± 0,0001c |
1,32 ± 0,08c |
0,11 ± 0,02c |
|
M3 |
1,38 ± 0,05b |
0,0032 ± 0,0006c |
1,06 ± 0,07c, d |
0,10 ± 0,01c |
|
M4 |
1,37 ± 0,05b |
0,0047 ± 0,0003a |
1,51 ± 0,06a |
0,18 ± 0,09b |
|
M5 |
1,40 ± 0,40b |
0,0040 ± 0,0001b |
0,81± 0,04d |
0,10 ± 0,01c |
|
M6 |
1,30 ± 0,02c |
0,0040 ± 0,0005b |
1,32 ± 0,06c |
0,107 ± 0,005c |
|
*Los datos se expresaron como media ± desviación estándar de los valores obtenidos por triplicado de los Experimentos. Las letras en cada columna indican diferencias significativas. |
||||
En la prueba de Salkowski, todas las cepas presentaron coloración naranja-rojiza en presencia de L-Trp, indicando la producción de compuestos indólicos) datos no incluidos en este documento). En los resultados de la detección y cuantificación de estos compuestos observamos que la muestra M2 produjo una concentración significativamente mayor de AIA en comparación con las demás cepas, con valores de 16,75µg/mL en presencia de L-Trp y 9,96µg/mL en ausencia de este aminoácido. Esta fue la única cepa que produjo indol en un medio sin suplementación del L-Trp, con una concentración de 0,13µg/mL. Por su parte, la cepa M4 también mostró una producción significativa de AIA, con una concentración de 4,57µg/mL en presencia de L-Trp. Además, detectamos la producción de indol en todas las cepas excepto la M1, especialmente en presencia de L-Trp. La M4, al igual que el control positivo presenta concentraciones de AIA e indol, ambos en presencia de L-Trp únicamente, con valores de 4,57µg/mL y 5,53µg/mL respectivamente. Ninguna de las cepas cuantificó concentraciones detectables de AIB (Tabla 3).
TABLA 3
Cuantificación de auxinas producidas por cinco cepas bacterianas aisladas de bioles
|
|
Concentración AIA (µg/mL) |
Concentración indol (µg/mL) |
Concentración AIB (µg/mL) |
|||
|
Código |
MRS |
MRS + L- triptofano |
MRS |
MRS + L- triptofano |
MRS |
MRS + L-triptofano |
|
Control (-) |
nd |
nd |
nd |
Nd |
nd |
nd |
|
M1 |
nd |
0,62 |
nd |
Nd |
nd |
nd |
|
M2 |
9,96 |
16,75 |
0,13 |
Nd |
nd |
nd |
|
M3 |
nd |
nd |
nd |
3,51 |
nd |
nd |
|
M4 |
nd |
4,57 |
nd |
5,53 |
nd |
nd |
|
M5 |
nd |
nd |
nd |
1,27 |
nd |
nd |
|
Control (+) |
nd |
4,41 |
nd |
0,21 |
nd |
nd |
|
nd: no detectable |
||||||
DISCUSIÓN
La clasificación de los géneros de las cepas de BAL que aislamos en este estudio, es reciente, por lo que, de estos géneros específicos aún no se reportan aplicaciones agrícolas específicas. Sin embargo, existen estudios previos que sugieren el potencial agrícola de bacterias del género Lactobacillus sp. Por ejemplo, encontramos reportes que algunas especies pueden actuar como promotoras del crecimiento vegetal al producir compuestos como la auxina AIA y participar en la solubilización de minerales (Mohite, 2013; Shrestha et al., 2014; Giassi et al., 2016; Fernández, 2022; Raman et al., 2022). En particular, investigaciones han demostrado que Lactobacillus paracasei, puede promover el crecimiento de plantas, como observamos en el estudio de Murthy et al., (2013), donde inocularon el suelo alrededor de las raíces de plantas de tomate con L. paracasei, las cuales mostraron un significativo aumento en el crecimiento en comparación con las plantas no tratadas. Específicamente se observó un aumento en la altura de las plantas, un incremento en el diámetro del tallo y un mayor número de hojas en las plantas tratadas. Además, encontraron que las plantas tratadas tenían un sistema radicular más desarrollado, una mayor capacidad para absorber nutrientes del suelo y tenían niveles más altos de auxinas, en comparación con las plantas no tratadas con L. paracasei.
A pesar de que existen pocos informes sobre las BAL en agricultura, estos hallazgos y los resultados que obtuvimos en este estudio respaldan la idea de que las BAL, como L. paracasei, pueden desempeñar un papel importante como herramientas para mejorar la productividad agrícola de manera sostenible. Además, cuando comparamos nuestros datos de clasificación molecular con estudios previos, como el realizado por WingChing et al. (2021), demuestran la consistencia de nuestros resultados y confirman la eficacia de las técnicas de identificación molecular seleccionadas para la detección de cepas BAL. Esta validación fortalece la relevancia de nuestra investigación y resalta su contribución al campo de la agricultura sostenible y la biotecnología agrícola.
Los resultados que obtuvimos de las cinéticas de crecimiento muestran variaciones significativas en la capacidad de las cepas aisladas para crecer en diferentes condiciones de cultivo. Al comparar nuestros datos con investigaciones previas, encontramos similitudes en los valores de DO máximo obtenidos, lo que sugiere que existe una coherencia con el comportamiento de las cepas bajo diferentes condiciones. Por ejemplo, Fernández (2022) reportó valores de DO máximo de 1,536 ± 0,195 y 1,629 ± 0,229 para dos aislamientos de L. paracasei obtenidos de residuos agroindustriales, lo cual es consistente con nuestros resultados. Además, Nguyen-Sy et al. (2020) informaron un DO máximo de 2,0 para una cepa de L. paracasei extraída de soya fermentada, lo que sugiere una amplia variabilidad en el crecimiento entre diferentes cepas y condiciones de crecimiento. En ambos estudios revelaron valores de DO máximo consistentes con los obtenidos en nuestra investigación, lo que valida la robustez de nuestros datos de crecimiento bacteriano.
Destacamos las discrepancias en los valores de DO máximo y tasas de crecimiento entre las cepas estudiadas, particularmente en relación con la presencia o ausencia de L-Trp en el medio de cultivo. Por ejemplo, observamos que la cepa M2 mostró una producción intermedia de AIA y una tasa de crecimiento bacteriano consistente tanto en presencia o en ausencia de L-Trp, lo que sugiere una adaptación robusta a diferentes condiciones nutricionales. Por otro lado, la cepa M5 exhibió una menor producción de AIA y una tasa de crecimiento más lenta en presencia de L-Trp, lo que indica una posible dependencia de esta cepa en la presencia de este aminoácido para su crecimiento óptimo. Estas variaciones en la respuesta de las cepas al L-Trp las podemos atribuir a diferencias en su metabolismo, a la fuente de aislamiento y a su capacidad para sintetizar compuestos indólicos (Suliasih & Widawati, 2020; Fernández, 2022).
Basándonos en los resultados que obtuvimos por la primera prueba de caracterización química, donde todas las cepas que estudiamos presentan resultados positivos para la prueba de Salkowski en presencia de L-Trp, podemos confirmar estos datos según lo expuesto Bric et al., (1991). Este estudio indica que la presencia de compuestos indólicos está directamente relacionada con la intensidad del color rojo o naranja producido, que surge de una reacción oxidativa a partir de ácido sulfúrico, donde mediante una transaminación del grupo amino del indol por el cloro del FeCl3. Además, identificamos cinco compuestos que generan una reacción visible mediante cambio de color en presencia de Trp: indol, 5-hidroxi-indolacético, indol acetamida, ácido indolacético y triptamina (Bric et al., 1991; Ahmad et al., 2005). Lo que sugieren que las cepas estudiadas en esta investigación pueden producir alguno de estos compuestos indólicos. Sin embargo, dado que esta prueba es cualitativa, se recomienda la aplicación de técnicas analíticas más precisas, como HPLC o MS/MS, para determinar la concentración exacta de estos compuestos (Bric et al., 1991; Mantilla, 2007; Mohite, 2013; Abad, 2021). Un ejemplo que respalda estos resultados es el estudio realizado por Mohite, (2013), quién aisló dos cepas identificadas como Lactobacillus casei y Bacillus cereus de muestras de rizosfera de plátano, rizosfera de trigo y en la rizosfera del maíz. Estas cepas mostraron un cambio de color durante la prueba de Salkowski, y posteriormente, al ser cuantificadas mediante una técnica analítica, se detectaron concentraciones de AIA de 50 y 60µg/mL, respectivamente. Al relacionar los datos de cuantificación de compuestos indólicos con las cinéticas de crecimiento, observamos que la cepa M2 en presencia de L-Trp produce la mayor cantidad de AIA, pero la biomasa producida disminuye. Esto sugiere que la producción de AIA no está directamente relacionada con la biomasa bacteriana, sino más bien con la presencia de L-Trp como promotor de la producción de compuestos indólicos. Además, al analizar esta cepa en ausencia de L-Trp, observamos una producción de AIA menor pero una biomasa superior, por lo que sugerimos que esta cepa puede ser podría ser una candidata promisoria para el desarrollo de bioinsumos agrícolas basados en microorganismos promotores del crecimiento vegetal. Dadas las diferencias significativas en las concentraciones y tipos de compuestos indólicos producidos por las cepas M1, M4 y M5, a pesar de su identificación molecular como Lacticaseibacillus paracasei, observamos diferentes comportamientos en su caracterización, ¿por? lo que sugerimos realizar análisis posteriores de identificación y caracterización para una correcta clasificación. Por ejemplo, la cepa M1 muestra una tasa de crecimiento específica más alta en presencia de L-Trp que en ausencia del mismo. Sin embargo, esta cepa no produce AIA en concentraciones detectables, según los resultados de la cuantificación de compuestos indólicos. Esto sugiere que el aumento en la tasa de crecimiento específica podría estar relacionado con otros factores que no están directamente vinculados con la presencia del aminoácido Trp o a la producción de AIA.
Por otra parte, la cepa M4 exhibe una alta tasa de crecimiento específica en ambos medios, sin L-Trp y con L-Trp. Además, esta cepa muestra una producción significativa de AIA e indol en presencia de L-Trp, como se observa en los resultados que obtuvimos en esta investigación. Esto indica una posible relación entre la alta tasa de crecimiento específica y la capacidad de producir compuestos indólicos en esta cepa. En cuanto a la cepa M5, muestra una tasa de crecimiento específica más baja en medio suplementado con L-Trp que en medio sin suplementar. en comparación con MRS sin L-triptófano. Además, esta cepa no produce AIA en concentraciones detectables en ninguno de los medios. Esto sugiere que la baja producción de AIA puede estar asociada con la disminución de la tasa de crecimiento específica en presencia de L-Trp.
Estas variaciones que observamos en la respuesta al L-Trp entre las cepas estudiadas podría estar relacionado con diferencias en la capacidad metabólica, la disponibilidad nutricional, la actividad enzimática y la regulación génica de la síntesis de compuestos indólicos a partir de la ruta del Trp (Keswani et al., 2020; Mora‐Villalobos et al., 2020). Por ejemplo, M2 podría tener una capacidad metabólica más robusta para sintetizar AIA a partir de indol-3-piruvato, independientemente de la disponibilidad de L-Trp en el medio, mientras la cepa M5, que exhibió una menor producción de AIA y una tasa de crecimiento más lenta en presencia de L-triptófano, lo podríamos atribuir a esta cepa se ve favorecida por una vía metabólica independiente del Trp (Keswani et al., 2020).
Por otra parte, al comparar nuestras concentraciones de AIA con estudios previos, por ejemplo, Turaeva et al., (2022), quién reportó una producción de 6,175mg/mL de AIA por parte de una cepa asilada de Lactobacillus plantarum, por su parte, Panetto et al., (2023) reportó una producción de AIA en una concentración de 7,25µg/mL en una cepa de L. acidophilus, comparando estos resultados con los obtenidos por la cepa M2, en presencia y ausencia de L-Trp lo que confirma a la cepa M2 como una cepa promisoria para aplicación agrícola. En la literatura existen reportes de muchas BPCP que por inducción de L-Trp generan rendimientos inclusive mayores a los reportados por la M2, pero son otras especies de bacterias, por ejemplo, especies de Bacillus como lo muestra Giassi et al., (2016), donde se reportan concentraciones hasta de 21,07µg/mL. Esto respalda la idea de que nuestras cepas podrían tener potencial como bioinsumos agrícolas, especialmente considerando su capacidad para producir compuestos indólicos en diferentes condiciones nutricionales.
En conclusión, esta investigación ha arrojado luz sobre la relación entre la producción de compuestos indólicos, específicamente AIA e indol, y las cinéticas de crecimiento de las cepas bacterianas estudiadas. Hemos demostrado que la presencia de L-Trp en el medio de cultivo puede influir significativamente en la producción de AIA, así como en el crecimiento bacteriano, lo que sugiere una interconexión entre el metabolismo de los compuestos indólicos y la viabilidad celular. Además, hemos identificado cepas prometedoras, como la M2, que muestran un potencial significativo como bioinsumos agrícolas para promover el crecimiento vegetal y mejorar la productividad de los cultivos de manera sostenible. Estos hallazgos no solo amplían nuestra comprensión de los mecanismos de síntesis de AIA en bacterias promotoras del crecimiento vegetal, sino que también ofrecen una base sólida para futuras investigaciones en el campo de la agricultura sostenible y la biotecnología agrícola. En última instancia, nuestro estudio contribuye al desarrollo de prácticas agrícolas más eficientes y respetuosas con el medio ambiente, con el potencial de beneficiar tanto a los agricultores como al medio ambiente. Para futuras investigaciones, recomendamos continuar explorando más a fondo la actividad funcional de los compuestos indólicos producidos por las cepas estudiadas, y realizar estudios adicionales para evaluar su efectividad como bioinsumos agrícolas en condiciones de campo. Esto incluye investigaciones sobre sus mecanismos de acción, su interacción con las plantas y su capacidad para mejorar la productividad de los cultivos en entornos agrícolas reales. Estos esfuerzos contribuirán a maximizar el potencial de nuestras cepas como herramientas para una agricultura más sostenible y productiva.
AGRADECIMIENTOS
Agradecemos al Laboratorio CENIBiot-CeNAT por permitir el desarrollo de la investigación en sus instalaciones, al Instituto Tecnológico de Costa Rica, por la beca suministrada para el desarrollo de la investigación, así como a todos los investigadores involucrados en el desarrollo de este estudio. Los resultados de este trabajo son parte del proyecto FI-041B-19, financiado por el Ministerio de Ciencia, Innovación, Tecnología y Telecomunicaciones de Costa Rica.
ÉTICA, CONFLICTO DE INTERESES Y DECLARACIÓN DE FINANCIAMIENTO
Declaramos haber cumplido con todos los requisitos éticos y legales pertinentes, tanto durante el estudio como en la preparación de este documento; que no hay conflictos de interés de ningún tipo, y que todas las fuentes financieras se detallan plena y claramente en la sección de agradecimientos. Asimismo, estoy/estamos de acuerdo con la versión editada final de esta publicación. El respectivo documento legal firmado se encuentra en los archivos de la revista.
La declaración de contribución de cada autor es la siguiente: K.M.C.: Desarrollo del trabajo experimental de todo el estudio, análisis de datos y preparación del manuscrito. J.M.Z.: Diseño de método de aislamiento y cinéticas de crecimiento. M.C.P.: Diseño y análisis de datos de cuantificación de fitohormonas producidas por las BAL aisladas. C.O.O.: Diseño experimental del proceso de extracción de rRNA para sus análisis por 16S rRNA y MLST. E.A.V.: Diseño del estudio general, guía en el análisis de datos experimentales, acompañamiento durante la investigación. Todos los coautores: preparación y aprobación final del manuscrito.
REFERENCIAS
Abad, M. E. (2021). Aislamiento e identificación de bacterias endófitas productoras de ácido indolacético a partir de plantas de lenteja de agua del género Spirodela. [Tesis de Ingeniería, Universidad de las Fuerzas Armadas]. https://repositorio.catie.ac.cr/handle/11554/3123
Ahmad, F., Ahmad, I., & Khan, M. S. (2005). Indole acetic acid production by the indigenous isolates of Azotobacter and fluorescent Pseudomonas in the presence and absence of tryptophan. Turkish Journal of Biology, 29(1), 29-34. https://journals.tubitak.gov.tr/biology/vol29/iss1/5
Altschul, S. F., Gish, W., Miller, W., Myers, E. W., & Lipman, D. J. (1990). Basic local alignment search tool. Journal of Molecular Biology, 215(3), 403-410. https://doi.org/10.1016/S0022-2836(05)80360-2
Borris, R., Fan, B., Wang, C., Song, X., Ding, X, Wu, L., Wu, H., Gao, X. (2018). Bacillus velezensis FZB42 in 2018: The gram-positive model strain for plant growth promotion and biocontrol. Frontier, Microbiology, 9, 2491. https://doi.org/10.3389/fmicb.2018.02491
Bric, J., Bostock, R. & Silverstone, S. (1991). Rapid in Situ assay for indoleacetic acid production by bacteria immobilized on a nitrocellulose membrane. Applied environmental microbiology, 57(2), 535-538. https://tinyurl.com/22kxzrz3
Castillo, G., Altuna, B., Michelena, G., Sánchez-Bravo, J., Acosta, M., & Acosta, C. M. (2005). Cuantificación del contenido de ácido indolacético (AIA) en un caldo de fermentación microbiana. Anales de Biología, 27, 137-142.
Compant, S., Clément, C., & Sessitsch, A. (2010). Plant growth-promoting bacteria in the rhizo- and endosphere of plants: Their role, colonization, mechanisms involved and prospects for utilization. Soil Biology and Biochemistry, 42(5), 669–678. https://doi.org/10.1016/j.soilbio.2009.11.024
Dong, C., Wang, G., Du, M., Niu, C., Zhang, P., Zhang, X., Ma, D., Ma, F., & Bao, Z. (2020). Biostimulants promote plant vigor of tomato and strawberry after transplanting. Scientia Horticulturae, 267, 109355. https://doi.org/10.1016/j.scienta.2020.109355
du Jardin, P. (2015). Plant biostimulants: Definition, concept, main categories and regulation. Scientia Horticulturae, 196, 3–14. https://doi.org/10.1016/j.scienta.2015.09.021
Ehman, A. (1977). The Van URK-Salkowski reagent — a sensitive and specific chromogenic reagent for silica gel thin-layer chromatographic detection and identification of indole derivatives. Journal of Chromatography A, 132(7746), 267–276.
Fernández. S. (2022). Caracterización de bacterias ácido lácticas (BAL) para la producción de ácido láctico (AL) a escala de planta piloto. [Tesis de Licenciatura, Universidad de Costa Rica]. https://tinyurl.com/2bjsw7e9
Fonseca-Carreño, N. E., Salamanca-Merchan, J. D., & Vega-Baquero, Z. Y. (2019). La agricultura familiar agroecológica, una estrategia de desarrollo rural incluyente. Una revisión. Revista Temas Agrarios, 24(2), 96–107. https://doi.org/10.21897/rta.v24i2.1356
Giassi, V., Kiritani, C., & Kupper, K. C. (2016). Bacteria as growth-promoting agents for citrus rootstocks. Microbiological Research, 190, 46–54. https://doi.org/10.1016/j.micres.2015.12.006
Glick, B. R. (2012). Plant Growth-Promoting Bacteria: Mechanisms and Applications. Scientifica, 1–15. https://doi.org/10.6064/2012/963401
Goswami, M., & Deka, S. (2020). Plant growth-promoting rhizobacteria alleviators of abiotic stresses in soil: A review. Pedosphere, 30(1), 40-61. https://doi.org/10.1016/S1002-0160(19)60839-8
Grover, M., Ali, S. Z., Sandhya, V., Rasul, A., & Venkateswarlu, B. (2011). Role of microorganisms in adaptation of agriculture crops to abiotic stresses. World Journal of Microbiology and Biotechnology, 27(5), 1231–1240. https://doi.org/10.1007/s11274-010-0572-7
Guerrero, J. G., (2022). Efectos del biol en el cultivo de melón (Cucumis melo L.) y su impacto en el rendimiento. [Tesis de Ingeniería, Universidad Técnica de Babahoyo]. http://dspace.utb.edu.ec/handle/49000/13284
Hall, T. A. (1999) BioEdit: A user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucleic Acids Symposium Series, 41, 95-98.
Kang, S. M., Radhakrishnan, R., You, Y. H., Khan, A. L., Park, J. M., Lee, S. M., & Lee, I. J. (2015). Cucumber performance is improved by inoculation with plant growth-promoting microorganisms. Acta Agriculture Scandinavica Section B: Soil and Plant Science, 65(1), 36–44. https://doi.org/10.1080/09064710.2014.960889
Khalid, A., Arshad, M., & Zahir, Z. A. (2004). Screening plant growth-promoting rhizobacteria for improving growth and yield of wheat. Journal of Applied Microbiology, 96(3), 473–480. https://doi.org/10.1046/j.1365-2672.2003.02161.x
Keswani, C., Singh, S. P., Cueto, L., García-Estrada, C., Mezaache-Aichour, S., Glare, T. R., Borriss, R., Singh, S. P., Blázquez, M. A., & Sansinenea, E. (2020). Auxins of microbial origin and their use in agriculture. Applied Microbiology and Biotechnology, 104(20), 549–8565. https://doi.org/10.1007/s00253-020-10890-8
Linares-Gabriel, A., López-Collado, C. J., Tinoco-Alfaro, C. A., Velasco-Velasco, J., & López-Romero, G. (2017). Application of biol, inorganic fertilizer and superabsorbent polymers in the growth of heliconia (Heliconia psittacorum cv. Tropica). Revista Chapingo Serie Horticultura, 23(1), 35–48. https://doi.org/10.5154/r.rchsh.2016.02.004
Mantilla, M. E. (2007). Evaluación de la acción de un bioinoculante sobre un cultivo de crisantemo (Chrysabthemum morifolium var. yoko ono) en periodo de enraizamiento. [Tesis Grado, Pontificia Universidad Javeriana]. https://repository.javeriana.edu.co/handle/10554/8381
Mazid, M., & Khan, T. A. (2014). Future of Bio-fertilizers in Indian Agriculture: An Overview. International Journal of Agricultural and Food Research, 3(3), 10–23. https://doi.org/10.24102/ijafr.v3i3.132
Mohite, B. (2013). Isolation and characterization of indole acetic acid (IAA) producing bacteria from rhizospheric soil and its effect on plant growth. Journal of Soil Science and Plant Nutrition, 13(3), 638–649. https://doi.org/10.4067/S0718-95162013005000051
Montero-Zamora, J., Cortés-Muñoz, M., Esquivel, P., Mora-Villalobos, J. A., & Velázquez, C. (2020). Growth conditions and survival kinetics during storage of Lactobacillus rhamnosus GG for the design of a sustainable probiotic whey-based beverage containing Costa Rican guava fruit pulp. Journal of Food Science, 85(10), 3478–3486. https://doi.org/10.1111/1750-3841.15430
Mora‐Villalobos, J. A., Montero‐Zamora, J., Barboza, N., Rojas‐Garbanzo, C., Usaga, J., Redondo‐Solano, M., Schroedter, L., Olszewska‐Widdrat, A., & López‐Gómez, J. P. (2020). Multi‐product lactic acid bacteria fermentations: A review. Fermentation, 6(1), 23. https://doi.org/10.3390/fermentation6010023
Muñiz, C. A., (2023). Beneficios del biol en el cultivo de pepino (Cucumis sativus). [Tesis de Ingeniería, Universidad Técnica de Babahoyo]. http://dspace.utb.edu.ec/handle/49000/14100?show=full
Murthy, K. N., Malini, M., Savitha, J., & Srinivas, C., (2013). Lactic acid bacteria (LAB) as plant growth promoting bacteria (PGPB) for the control of wilt of tomato caused by Ralstonia solanacearum. Pest Manag. Hort. Ecosyst. 18, 60–65.
Naik, K., Mishra, S., Srichandan, H., Singh, P. K., & Sarangi, P. K. (2019). Plant growth promoting microbes: Potential link to sustainable agriculture and environment. Biocatalysis and Agricultural Biotechnology, 21, 101326. https://doi.org/10.1016/j.bcab.2019.101326
Nguyen-Sy, T., Yew, G. Y., Chew, K. W., Nguyen, T. D. P., Tran, T. N. T., & Le, T. D. H. (2020). Potential cultivation of Lactobacillus pentosus from human breastmilk with rapid monitoring through the spectrophotometer method. Processes, 8(8), 1–9. https://doi.org/10.3390/pr8080902
Panetto, L. D., Doria, J., Santos, C. H. B., Frezarin, E. T., Sales, L. R., de Andrade, L. A., & Rigobelo, E. C. (2023). Lactic bacteria with Plant-Growth-Promoting properties in potato. Microbiology Research, 14(1), 279–288. https://doi.org/10.3390/microbiolres14010022
Rahmoune, B., Morsli, A., Khelifi-Slaoui, M., Khelifi, L., Strueh, A., Erban, A., Kopka, J., Prell, J., & Van Dongen, J. T. (2017). Isolation and characterization of three new PGPR and their effects on the growth of Arabidopsis and Datura plants. Journal of Plant Interactions, 12(1), 1–6. https://doi.org/10.1080/17429145.2016.1269215
Raman, J., Kim, J., Choi. K. R., Eun, H., Yang, D., Ko, Y., & Kim, S. (2022). Application of Lactic Acid Bacteria (LAB) in sustainable agriculture: Advantages and limitations. International Journal of Molecular Sciences, 23(14), 7784. https://doi.org/10.3390/ijms23147784.
Restrepo, J. (2007). Manual práctico. El A, B, C de la agricultura orgánica y harina de rocas. Printex. https://tinyurl.com/2b7jscgr
Shrestha, A., Kim, B. S., & Park, D. H. (2014). Biocontrol science and technology biological control of bacterial spot disease and plant growth-promoting effects of lactic acid bacteria on pepper. Biocontrol Science and Technology, 37–41. https://doi.org/10.1080/09583157.2014.894495
Solís-Oba, M. M., Castro-Rivera, R., Villegas-Luna, A., Cruz-Murillo, A., Solís-Oba, A., Castro-Ramos, J. J., Romero-Rodríguez, A., Juárez-Rangel, A. P., Pacheco-Ortiz, J. A., & Aguilar-Benítez, G. (2021). Evaluación de biol, bocashi, composta y vermicomposta en las variables morfológicas del cultivo de espinaca (Spinacia oleracea L.). Brazilian Journal of Animal and Environmental Research, 4(3), 3649–3662. https://doi.org/10.34188/bjaerv4n3-070
Suliasih, & Widawati, S. (2020). Isolation of indole acetic acid (IAA) producing Bacillus siamensis from peat and optimization of the culture conditions for maximum IAA production. IOP Conference Series: Earth and Environmental Science, 572(1). https://doi.org/10.1088/1755-1315/572/1/012025
Szkop, M., & Bielawski, W. (2013) A simple method for simultaneous RP-HPLC. Determination of indolic compounds related to bacterial biosynthesis of indole-3-acetic acid. Antonie van Leeuwenhoek, 103, 683-691. https://doi.org/10.1007/s10482-012-9838-4
Tamura, K., Stecher, G., & Kumar, S. (2021) MEGA11: Molecular Evolutionary Genetics Analysis version 11. Molecular Biology and Evolution 38, 3022-3027. https://doi.org/10.1093/molbev/msab120
Turaeva, B. I., Fo qizi, K. K., Soliev, A. B., y Kutlieva, G. J. (2022). Gibberellic and indole acetic acids producing features of bacteria from the genus Lactobacillus and their effect on plant development. Asian Journal of Biological and Life Sciences, 10(3), 681–686. https://tinyurl.com/2b2wh9sl
WingChing, R., Redondo, M., Usaga, J., Uribe, L., & Barboza, N. (2021). Tipificación con secuencias multilocus en Lactobacillus casei procedentes de ensilados de cáscara de piña. Agronomía Mesoamericana, 32(2), 508–522. https://doi.org/10.15517/am.v32i2.42182
Xiu, P. (2018). Efectos de bioles en brócoli (Brassica oleracea) y lechuga (Lactuca sativa) en la zona hortícola de Cartago, Costa Rica. [Tesis de Maestría, Centro Agronómico Tropical de Investigación y Enseñanza]. Repositorio CATIE.
Zagoya, J. (2013) Evaluación de biofertilizantes y factores para su innovación con productores de maíz en San Felipe Teotlalcingo, Puebla. [Tesis de Maestría en Ciencias. Instituto de Enseñanza e Investigación en Ciencias Agrícolas, Campus Puebla (COLPOS)]. http://colposdigital.colpos.mx:8080/xmlui/handle/10521/2206
Zanabria, J. I. (2019). Evaluación de la calidad de biol de segunda y tercera generación de estiércol de cuy producido en un biodigestor instalado en el instituto regional de la costa de la UNALM. [Tesis de Ingeniería, Universidad Nacional Agraria]. https://tinyurl.com/23hhjy8n
Zúñiga. O. M. (2009). Crecimiento de Bacillus pumilus productor de la auxina ácido indolacético, como base para formular biofertilizante en polvo. [Tesis de Ingeniería, Universidad Austral de Chile]. http://colposdigital.colpos.mx:8080/xmlui/handle/10521/2206
