
Bacterias cultivables resistentes a oxitetraciclina durante el proceso de vermicompostaje de excretas bovinas
Lorena Uribe-Lorío1,3![]() , Rodolfo WingChing-Jones2,5
, Rodolfo WingChing-Jones2,5![]() , Daniela Vidaurre-Barahona3
, Daniela Vidaurre-Barahona3![]() , Lidieth Uribe1,4
, Lidieth Uribe1,4![]() & Luis Felipe Aráuz1
& Luis Felipe Aráuz1![]()
1. Universidad de Costa Rica, Escuela de Agronomía, San Pedro, Costa Rica; lorena.uribe@ucr.ac.cr, lidieth.uribe@ucr.ac.cr; felipe.arauz@ucr.ac.cr,
2. Universidad de Costa Rica, Escuela de Zootecnia, San Pedro, Costa Rica; rodolfo.wingching@ucr.ac.cr
3. Universidad de Costa Rica, Centro de Investigación en Biología Celular y Molecular (CIBCM), San Pedro, Costa Rica; danividaurre@gmail.com
4. Universidad de Costa Rica, Centro de Investigaciones Agronómicas (CIA), San Pedro, Costa Rica.
5. Universidad de Costa Rica, Centro de Investigación en Nutrición Animal, San Pedro, Costa Rica.
Recibido 15-I-2023 □ Corregido 23-III-2023 □ Aceptado 25-IV-2023
DOI: https://doi.org/10.22458/urj.v15i1.4586
|
ABSTRACT. “Culturable bacteria resistant to oxytetracycline during the vermicomposting of bovine excreta”. Introduction: Bacteria with resistance genes to the antibiotic oxytetracycline have been detected in dairy cow manure worldwide, however, it is common a practice to use this manure as fertilizer. Vermicomposting can reduce the problem, but this subject has not been evaluated in Costa Rica. Objective: To analyze the presence of bacteria resistant to oxytetracycline during vermicomposting in a Costa Rican dairy farm. Methods: We inoculated broths supplemented with oxytetracycline from fresh manure, precomposted manure, vermicompost and "compost teas" with and without molasses. We prepared “compost teas” with and without molasses from this sample. We extracted DNA from these liquid cultures and mass-sequenced the 16S ribosomal amplicon. Results: We classified 105 292 sequences in 58 amplicon sequence variants from broths supplemented with oxytetracycline, most of them identified as Proteobacteria, Bacteroidetes and Firmicutes. Fresh manure had the most resistant bacteria (32), followed by the “teas” without and with molasses. Precomposting and vermicomposting decreased the number and type of resistant bacteria. However, preparation of the teas caused the multiplication of bacterial genera recognized for their ability to accumulate resistance determinants. Conclusion: Precomposting and vermicomposting decreased the number and type of resistant bacteria.
Key words: antimicrobials, tetracycline, bovine manure, lombricompost, lombricompost tea.
|
RESUMEN. Introducción: A nivel mundial se han detectado bacterias con genes de resistencia al antibiótico oxitetraciclina en estiércol de vacas lecheras, sin embargo, es una práctica común utilizar este estiércol como fertilizante. El vermicompostaje puede reducir el problema, pero este tema no ha sido evaluado en Costa Rica. Objetivo: Analizar la presencia de bacterias resistentes a la oxitetraciclina durante el vermicompostaje en una finca lechera costarricense. Métodos: A partir de estiércol fresco, precompostado, lombricomposta, y tés de composta con y sin melaza y tés de composta con y sin melaza, inoculamos caldos suplementados con oxitetraciclina. Extrajimos el ADN de estos cultivos líquidos y secuenciamos en masa el amplicón ribosómico 16S. Resultados: Clasificamos 105 292 secuencias en 58 variantes de secuencia de amplicón de caldos suplementados con oxitetraciclina, la mayoría identificadas como Proteobacteria, Bacteroidetes y Firmicutes. El estiércol fresco tuvo más bacterias resistentes (32), seguido de los “tés” sin y con melaza. El precompostaje y el vermicompostaje disminuyeron la cantidad y variedad de bacterias resistentes. Sin embargo, la preparación de los tés provocó la multiplicación de géneros bacterianos reconocidos por su capacidad de acumular determinantes de resistencia. Conclusión: El precompostaje y el vermicompostaje disminuyeron el número y tipo de bacterias resistentes.
Palabras clave: antimicrobianos, tetraciclina, estiércol bovino, lombricompost, té de lombricompost.
|
El uso de excretas en la agricultura es una práctica difundida en los sistemas de producción que, si bien constituye un aporte de nutrientes a los agroecosistemas, puede introducir patógenos y bacterias resistentes a antibióticos (Pandey et al., 2018; Black et al., 2021). Una alternativa para la gestión de estos residuos es la producción de vermicompost y sus derivados como el “té de vermicompost” (Artavia et al., 2010; Gupta et al., 2016; Zamora et al., 2017). El vermicompostaje es una tecnología en la que se transforman los residuos orgánicos por acción de las lombrices en un abono orgánico (Kiyasudeen et al., 2016; Yatoo et al., 2021). Durante esta transformación y en el producto final, los microorganismos juegan un papel en la descomposición de los residuos (Romero-Tepal et al., 2014; Aira et al., 2016; Hu et al., 2020), en el control de plagas y enfermedades y en la estimulación del crecimiento de las plantas mediante la mineralización del fósforo y fijación de nitrógeno, entre otros (Yatoo et al. 2021).
En general, se considera esta práctica una de las mejores alternativas para el tratamiento de residuos contaminados con patógenos ya que este proceso provoca la bioconversión de residuos agrícolas, biomédicos y municipales en productos libres de enterobacterias (Eastman et al., 2001; Uribe et al., 2009; Pathma & Sakthivel, 2012), además de la degradación de antibióticos, como las tetraciclinas (Huang et al., 2018; Wang et al., 2019; Li et al., 2022).
A partir del vermicompost se puede obtener tés, un tipo de infusión que se prepara con vermicompost y agua, que son utilizados como fertilizantes líquidos para aplicar a nivel foliar o al suelo (Arancon et al., 2007; 2019; Pant et al., 2012). Durante este proceso se extraen los microorganismos y los nutrientes solubles del abono (Scheuerell & Mahaffee, 2004; Arancon et al., 2007; Fritz et al., 2012; Pane et al., 2014; Kiyasudeen et al., 2016; Zamora et al., 2017), a lo que se atribuye que el té retenga la capacidad del vermicompost para estimular el crecimiento de las plantas y el control de plagas y enfermedades de estas (Scheuerell & Mahaffee 2002; Arancon et al., 2019, Yatoo et al. 2021). Una práctica común consiste en la adición de azúcares como la melaza, al considerarse que el número de bacterias beneficiosas aumenta (Duffy et al., 2004; Kiyasudeen et al., 2016). Sin embargo, el Servicio de Investigación Agrícola del Departamento de Agricultura de Estados Unidos (USDA por sus siglas en inglés) y otros autores han informado que la adición de fuentes de C a los tés, inducen la proliferación de coliformes y patógenos bacterianos entéricos, como E. coli y Salmonella (Duffy et al., 2004; Giacomini, 2012; Kiyasudeen et al., 2016). Además, existe el riesgo potencial de la presencia de bacterias resistentes a antibióticos en los materiales derivados de diferentes tipos de estiércol que podrían ser transportados a suelos y plantas (Zalewska et al., 2021).
Los antibióticos se han utilizado en los sistemas de producción de animales de interés zootécnico durante más de 60 años para la prevención y terapia de patologías (Virto et al., 2022). A nivel mundial, la mayoría de los antibióticos que se utilizan en la producción de leche se dirigen a la promoción del crecimiento (cuando se permite), a la profilaxis y al tratamiento de enfermedades como la mastitis o la metritis, además del secado de la vaca (Zalewska et al., 2021). Dentro de los antimicrobianos más utilizados para controlar las enfermedades bacterianas en el ganado lechero en Estados Unidos están las tetraciclinas (Oliver et al., 2020). En Costa Rica, la tetraciclina como medicamento es el más utilizado en ganado bovino lechero, habiendo reportes de su uso en el 72% de 60 fincas artesanales de Costa Rica. El período de retiro aconsejado después del último tratamiento con este antimicrobiano es de 72 a 144 horas (Tortós et al., 2006; Carmona & Vindas, 2008).
Se han detectado tanto residuos de tetraciclinas como genes y bacterias resistentes a este antimicrobiano en excretas de ganado en diferentes estudios a nivel mundial (Casey et al., 2013; Kraemer et al., 2019; Oliver et al., 2020; Zalewska et al., 2021). Además, se ha estimado que la clortetraciclina presenta una tasa de excreción de 65% en excretas de ganado (Aust et al., 2008). La exposición de productores a bacterias resistentes es de gran preocupación, ya que puede trasladarse a las comunidades en riesgo por el contacto persona a persona (Silbergeld et al., 2008). Al respecto, se han detectado casos de infecciones con bacterias resistentes a meticilina en personas que viven y/o trabajan en granjas de producción porcina y bovina (Huijsdens et al., 2006; Carrel et al., 2014), por lo que la aplicación de estiércol en campos de cultivo cercanos a viviendas humanas puede aumentar el riesgo de infecciones resistentes a los antibióticos (Kumar et al., 2005). De hecho, la Organización de las Naciones Unidas para la Alimentación y Agricultura consideran al uso de antibióticos en el sector agropecuario un problema de salud pública, por lo que se ha desarrollado una estrategia global para la contención de la resistencia a los antibióticos, mediante recomendaciones para su uso en la agricultura (Organización de las Naciones Unidad para la Alimentación y la Agricultura [FAO], 2021).
En Costa Rica se ha detectado la presencia de genes de resistencia a oxitetraciclina en sedimentos cercanos a granjas porcinas (Vindas, 2019), en alimentos para cerdos y tilapias (Gutiérrez et al., 2010). En el caso de la leche bovina, se han reportado bacterias resistentes a tetraciclina y residuos de antimicrobianos de manera general (Calderón, 1992; Araya et al., 2005). El objetivo de esta investigación fue analizar la presencia de bacterias resistentes a la oxitetraciclina en las diferentes etapas del proceso de vermicompostaje de excretas bovinas en una explotación lechera, incluyendo los subproductos, mediante la secuenciación del amplicón 16S ribosomal.
MATERIALES Y MÉTODOS
Ubicación y muestreo: Realizamos el estudio durante el primer trimestre del año 2019, en el módulo lechero de la sede del Atlántico, Turrialba, Cartago, Costa Rica (660 metros sobre el nivel del mar) (9°54´13.63´´N 83°40´08.27´´O), en el Centro de Investigación en Biología Celular y Molecular (CIBCM) (9°56´23.13´´N 84°02´38.27´´O) y en el Centro de Investigaciones Agronómicas (CIA) (9°56´22.55´´N 84°02´43.25´´O), todos pertenecientes a la Universidad de Costa Rica. El módulo lechero es una finca dedicada a la producción de leche en condiciones tropicales, cuyo sistema manejaba 23 vacas en producción de la raza Jersey en el momento de la investigación.
Para efectos de este estudio, le dimos seguimiento a un lote de estiércol desde que ingresó al proceso de vermicompostaje hasta la elaboración de los tés de vermicompost. Colectamos muestras de las diferentes etapas: estiércol (CM-TB) día 0, estiércol precomposteado (CMC-TB) día 15, vermicompost (VC-TB) día 35. A partir del vermicompost preparamos el té de vermicompost (VCT-TB) y té de vermicompost con melaza (VCTM-TB) siguiendo la metodología descrita por Zamora et al., (2017). Para el té sin aditivo (VCT-TB), colocamos 100g del vermicompost en una bolsa de malla, la cual se sumergió en un recipiente de 10L con 5L de agua desionizada. Incubamos la mezcla con aireación constante durante 18 horas con una bomba de aire de 3L/min. Preparamos el té suplementado con melaza (VCTM-TB), utilizando el mismo procedimiento descrito anteriormente, añadiendo 10 ml de melaza. Terminado el proceso de preparación, se tomó muestras de ambos tés.
Recuperación de bacterias resistentes a oxitetraciclina: Para determinar la presencia de bacterias resistentes, inoculamos 1ml de muestra líquida (tés de vermicompost) y 0,1g de muestras sólidas (excretas y vermicompost) en 10ml de Caldo Nutritivo (Oxoid®) suplementado con 10μg/ml de oxitetraciclina (Sigma-Aldrich®) (Popowska et al., 2012). Debido a que no existen directrices para la determinación de la susceptibilidad para bacterias ambientales, los cortes para la determinación de la susceptibilidad/resistencia de un microorganismo se realizan con especies bacterianas de origen clínico, por lo que utilizamos la concentración de 10μg/ml basado en la recomendación para patógenos humanos entéricos, no entéricos y anaerobios según la guía CLSI (2017). Esta concentración es superior a la concentración mínima inhibitoria (CMI) intermedia para estos grupos bacterianos. Los tubos se incubaron por 72h o hasta que se presentó crecimiento a 30°C en condiciones aerobias.
Extracción de ADN: Centrifugamos 3ml del caldo de cultivo con crecimiento bacteriano y el botón fue sometido a extracción de ADN utilizando el kit de extracción de ADN Nucleospin (Macherey-Nagel®), según instrucciones del fabricante. La integridad del ADN de todas las muestras se examinó en geles de agarosa al 1% mediante electroforesis y se cuantificó utilizando el equipo NanoDrop 2000c Thermo Scientific® (Wilmington, DE, EE. UU). Los ácidos nucleicos se almacenarón a -20°C.
Secuencias: Para obtener los microbiomas que representan la comunidad bacteriana cultivable y resistente a tetraciclina de cada muestra, secuenciamos masivamente el ADN derivado de los cultivos en la compañía Macrogen Inc. (Corea del Sur), amplificando la región V3-V4 del gen 16S ARNr utilizando MiSeq Platform (Illumina. Inc), utilizando los imprimadores F: 5' TCGTCGGCAGCGTCAGATGTGTATAAGAGACAG 3' y R: 5' GTCTCGTGGGCTCGGAGATGTGTATAAGAGACAG 3'.
Procesamos los datos utilizando QIIME2 (versión 2018.11) (Bolyen et al., 2019) y sus complementos. La taxonomía de los ASV se asignó frente al conjunto de datos de referencia no redundante SILVA 16S/18S rDNA (SSURef 132 NR) (Quast et al., 2012) con el clasificador de características classify-sklearn. Esto dio origen a la tabla de ASVs, que permiten distinguir secuencias que se diferencian en un único nucleótido, lo que hace posible separar especies diferentes (Callahan et al., 2017). Comprobamos la clasificación taxonómica derivada del análisis bioinformático utilizando la base de datos curada de EzBioCloud (Yoon et al., 2017), que almacena secuencias del gen 16S rRNA de cepas tipo y de nombres válidamente publicados. Utilizamos los criterios publicados por Chun et al., (2010) para la asignación taxonómica de cada lectura: especie (x ≥ 97%), género (97%; x ≥ 94%), familia (94%; x ≥ 90%), orden (90%; x ≥ 85%), clase (85%; x ≥ 80%) y filo (80%; x ≥ 75%). Si se encontró una similitud por debajo del punto de corte, asignamos la lectura a un grupo "sin clasificar" (unc.).
Depositamos las secuencias crudas en la base de datos GenBank del NCBI bajo el bioproyecto PRJNA897901.
Análisis estadístico: Realizamos el análisis estadístico utilizando el software Plymouth Routines In Multivariate Ecological Research (PRIMER7) (Clarke & Goyle, 2015). Estimamos el índice de diversidad de Shannon (H') y Simpson de la matriz de ASVs y los valores de similitud de Bray-Curtis a partir de los datos con la transformación presencia/ausencia (debido a que las muestras son cultivos), de las abundancias de los ASV generados, así como de las sumas de las especies. La matriz de similitudes se formó utilizando la agrupación jerárquica aglomerativa y las divisiones resultantes se probaron con la rutina SIMPROF (perfiles de similitud) de tipo 1, para determinar similitudes significativas (p < 0,05) en la estructura de las comunidades de las muestras estudiadas. Visualizamos la matriz mediante una ordenación multidimensional no métrica (nMDS) para ilustrar las disimilitudes entre los agrupamientos de comunidades microbianas.
RESULTADOS
Bacterias resistentes: El análisis bioinformático de las comunidades bacterianas cultivables obtenidas a partir de los caldos suplementados con 10μg/ml de oxitetraciclina, dio como resultado 105 321 secuencias clasificadas en 58 ASVs (Tabla 1), la mayoría identificados en los filos Proteobacteria, Bacteroidetes y Firmicutes, de los cuáles las Proteobacterias y Bacteroidetes estuvieron en todas las muestras.
Además, se detectó un representante de Actinobacterias (Bifidobacterium) en CM-TB y uno de Spirochaetes (Treponema) en CMC-TB (Fig. 1).
Géneros bacterianos: Identificamos 32 géneros bacterianos (Tabla 2): 19 de ellos en la muestra de excretas frescas; los géneros fueron Bifidobacterium, Bacteroides, Succinivibrio, Alloprevotella, Faecalibacter, Streptococcus, Paeniclostridium, Turicibacter, Phascolarctobacterium, Succinivibrio, Citrobacter, y miembros no clasificados de Bacteroidaceae y Bacteroidales en los caldos suplementados con oxitetraciclina. La mayoría de estos géneros fueron exclusivos en CM-TB, y tuvieron solamente un representante en bajo número de secuencias (Tabla 1). Por su parte Aeromonas, Acinetobacter, Clostridium, Escherichia-Shigella y Proteus, se encontraron tanto en las excretas como en los tés de vermicompost. Escherichia ASV-2143 y A. colistiniresistens ASV-2213 se encontraron en mayor abundancia en CM-TB, además de Kurthia massiliensis ASV-830 (Tabla 1).
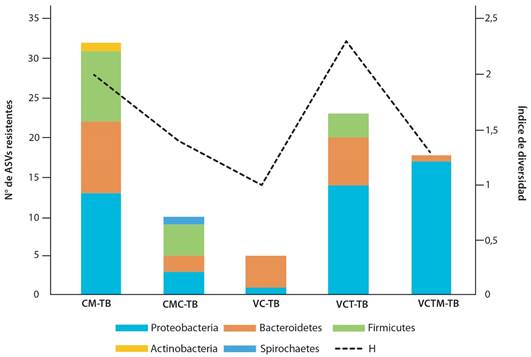
Fig. 1. Abundancia de filos presentes y diversidad de muestras de comunidades de bacterias cultivables resistentes a oxitetraciclina obtenidas de diferentes sustratos del proceso del vermicompostaje de excretas bovinas. Costa Rica, 2023.
TABLA 1
Identificación y abundancia de secuencias de ASVs crecidos en caldos suplementados con 10ug/ml de oxitetraciclina (TB) a partir de muestras del proceso de vermicompostaje. Costa Rica, 2023
|
ASV |
Phylum |
Especie |
CM-TB |
CMC-TB |
VC-TB |
VCT-TB |
VCTM-TB |
|
ASV-103 |
Actinobacteria |
Bifidobacterium pseudolongum |
41 |
0 |
0 |
0 |
0 |
|
ASV-179 |
Bacteroidetes |
Bacteroides graminisolvens |
42 |
0 |
0 |
0 |
0 |
|
ASV-182 |
Bacteroidetes |
unc. Bacteroidaceae |
68 |
0 |
0 |
0 |
0 |
|
ASV-194 |
Bacteroidetes |
unc. Bacteroidaceae |
35 |
0 |
0 |
0 |
0 |
|
ASV-217 |
Bacteroidetes |
unc. Bacteroidaceae |
44 |
0 |
0 |
0 |
0 |
|
ASV-258 |
Bacteroidetes |
unc. Muribaculaceae |
14 |
0 |
0 |
0 |
0 |
|
ASV-303 |
Bacteroidetes |
Alloprevotella sp. |
20 |
0 |
0 |
0 |
0 |
|
ASV-311 |
Bacteroidetes |
Prevotella paludivivens |
0 |
33 |
0 |
0 |
0 |
|
ASV-314 |
Bacteroidetes |
Prevotella sp. |
0 |
11 |
0 |
0 |
0 |
|
ASV-383 |
Bacteroidetes |
unc. Bacteroidales |
95 |
0 |
0 |
0 |
0 |
|
ASV-412 |
Bacteroidetes |
unc. Bacteroidales |
12 |
0 |
0 |
0 |
0 |
|
ASV-2673 |
Bacteroidetes |
Faecalibacter macacae |
37 |
0 |
0 |
0 |
0 |
|
ASV-2672 |
Bacteroidetes |
Myroides marinus |
0 |
0 |
27 629 |
0 |
0 |
|
ASV-592 |
Bacteroidetes |
Myroides marinus |
0 |
0 |
7 929 |
0 |
0 |
|
ASV-593 |
Bacteroidetes |
Myroides odoratus |
0 |
0 |
299 |
109 |
0 |
|
ASV-566 |
Bacteroidetes |
Chryseobacterium rhizoplanae |
0 |
0 |
70 |
19 |
189 |
|
ASV-493 |
Bacteroidetes |
unc. Saprospiraceae |
0 |
0 |
0 |
6 |
0 |
|
ASV-816 |
Firmicutes |
Bacillus sp. |
0 |
2 900 |
0 |
692 |
0 |
|
ASV-830 |
Firmicutes |
Kurthia massiliensis |
4 796 |
0 |
0 |
0 |
0 |
|
ASV-831 |
Firmicutes |
Kurthia sp. |
121 |
0 |
0 |
75 |
0 |
|
ASV-834 |
Firmicutes |
Rummeliibacillus pycnus |
0 |
122 |
0 |
0 |
0 |
|
ASV-835 |
Firmicutes |
Solibacillus silvestris |
0 |
1 552 |
0 |
114 |
0 |
|
ASV-841 |
Firmicutes |
Streptococcus sp. |
201 |
0 |
0 |
0 |
0 |
|
ASV-842 |
Firmicutes |
Streptococcus sp. |
25 |
0 |
0 |
0 |
0 |
|
ASV-918 |
Firmicutes |
Clostridium butyricum |
16 |
46 |
0 |
58 |
0 |
|
ASV-933 |
Firmicutes |
Clostridium senegalense |
0 |
0 |
0 |
73 |
0 |
|
ASV-936 |
Firmicutes |
Clostridium subterminale |
0 |
0 |
0 |
630 |
0 |
|
ASV-923 |
Firmicutes |
Clostridium neonatale |
32 |
0 |
0 |
0 |
0 |
|
ASV-1109 |
Firmicutes |
Paeniclostridium sordellii |
52 |
0 |
0 |
0 |
0 |
|
ASV-1423 |
Firmicutes |
Turicibacter sanguinis |
22 |
0 |
0 |
0 |
0 |
|
ASV-1457 |
Firmicutes |
Phascolarctobacterium sp. |
37 |
0 |
0 |
0 |
0 |
|
ASV-1930 |
Proteobacteria |
Comamonas thiooxydans |
0 |
0 |
9 514 |
0 |
0 |
|
ASV-2056 |
Proteobacteria |
Aeromonas caviae |
2 336 |
0 |
0 |
1 337 |
13 582 |
|
ASV-2057 |
Proteobacteria |
Aeromonas hydrophila |
0 |
0 |
0 |
1 064 |
616 |
|
ASV-2677 |
Proteobacteria |
Aeromonas hydrophila |
135 |
0 |
0 |
0 |
0 |
|
ASV-2071 |
Proteobacteria |
Aeromonas rivipollensis |
0 |
0 |
0 |
1 665 |
1 005 |
|
ASV-2675 |
Proteobacteria |
Aeromonas rivipollensis |
0 |
0 |
0 |
680 |
391 |
|
ASV-2674 |
Proteobacteria |
Aeromonas rivipollensis |
0 |
0 |
0 |
429 |
275 |
|
ASV-2678 |
Proteobacteria |
Aeromonas rivipollensis |
0 |
0 |
0 |
345 |
187 |
|
ASV-2676 |
Proteobacteria |
Aeromonas rivipollensis |
0 |
0 |
0 |
105 |
69 |
|
Proteobacteria |
2 406 |
0 |
0 |
5 505 |
7 513 |
||
|
ASV-2084 |
Proteobacteria |
Succinivibrio sp. |
31 |
0 |
0 |
0 |
0 |
|
ASV-2088 |
Proteobacteria |
Shewanella putrefaciens |
0 |
0 |
0 |
166 |
0 |
|
ASV-2137 |
Proteobacteria |
Citrobacter youngae |
37 |
0 |
0 |
0 |
0 |
|
Proteobacteria |
Escherichia-Shigella |
2 122 |
207 |
0 |
99 |
51 |
|
|
ASV-2146 |
Proteobacteria |
Proteus hauseri |
122 |
0 |
0 |
254 |
167 |
|
ASV-2209 |
Proteobacteria |
Acinetobacter baumannii |
0 |
74 |
0 |
0 |
75 |
|
ASV-2213 |
Proteobacteria |
Acinetobacter colistiniresistens |
1 092 |
0 |
0 |
878 |
223 |
|
ASV-2212 |
Proteobacteria |
Acinetobacter gandensis |
50 |
0 |
0 |
0 |
75 |
|
ASV-2199 |
Proteobacteria |
Acinetobacter indicus |
38 |
0 |
0 |
0 |
0 |
|
ASV-2202 |
Proteobacteria |
Acinetobacter johnsonii |
0 |
0 |
0 |
0 |
229 |
|
ASV-2680 |
Proteobacteria |
Acinetobacter lwoffii |
70 |
0 |
0 |
0 |
0 |
|
ASV-2204 |
Proteobacteria |
Acinetobacter schindleri |
155 |
0 |
0 |
0 |
32 |
|
ASV-2205 |
Proteobacteria |
Acinetobacter schindleri |
52 |
0 |
0 |
0 |
0 |
|
Proteobacteria |
0 |
1 064 |
0 |
219 |
81 |
||
|
ASV-2218 |
Proteobacteria |
Pseudomonas entomophila |
0 |
0 |
0 |
145 |
0 |
|
ASV-2228 |
Proteobacteria |
Pseudomonas hunanensis/alloputida |
0 |
0 |
0 |
0 |
39 |
|
ASV-2516 |
Spirochaetes |
Treponema sp. |
0 |
20 |
0 |
0 |
0 |
Observamos una disminución de los géneros con representantes resistentes en las excretas precomposteadas en comparación con las excretas frescas (19 vs 8), siendo los géneros Prevotella, Treponema y Rummeliibacillus exclusivos de CMC-TB.
En el vermicompost, solamente detectamos tres géneros (la menor cantidad del estudio): Myroides (3 ASVs), Comamonas (1 ASV) y Chryseobacterium (1 ASV), este último también presente en los tés de vermicompost.
Sin embargo, el proceso de elaboración de los tés aumentó el número de géneros con representantes resistentes a oxitetraciclina, ya que tanto en VC-TB como en VCTM-TB se identificaron 13 y 7 géneros respectivamente, comparado con los 3 del vermicompost. En ambas muestras detectamos una mayor cantidad de ASVs pertenecientes al género Aeromonas (8), seguido por Acinetobacter (2 en VCT-TB vs 7 en VCTM-TB). Aeromonas caviae ASV-2073 y A. sanarellii ASV-2056 fueron los ASVs más abundantes en estas muestras, y en menor medida en las excretas frescas. Otros géneros bacterianos compartidos entre VCT-TB y las muestras de excretas fueron Clostridium, Kurthia, Bacillus, Solibacillus, Proteus, Acinetobacter y Escherichia-Shigella. Estos tres últimos también fueron detectados en el cultivo de la muestra VCTM-TB, no obstante, ninguno de estos fue observado en el caldo inoculado con vermicompost.
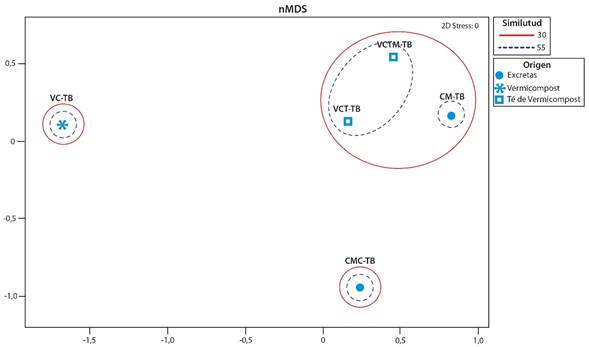
Fig. 2. Ordenación del escalamiento multidimensional no métrico (NMDS) basada en la matriz de distancia Bray-Curtis de comunidades bacterianas cultivables resistentes a 10ug/ml de oxitetraciclina del proceso de vermicompostaje de excretas bovinas. Costa Rica. 2023.
Mediante el análisis de escalamiento multidimensional no métrico, encontramos que las comunidades bacterianas identificadas en los caldos suplementados con oxitetraciclina de las excretas frescas los tés de vermicompost, tanto con o sin melaza, se agruparon con una similitud de Bray-Curtis de 30%, mientras que entre las muestras VCT-TB y VCTM-TB fue de 58% (p<0,05). No encontramos similitud entre las muestras de vermicompost sólido y excretas precompostadas, ni con ninguna de las otras muestras con respecto a sus comunidades bacterianas (Fig. 2).
TABLA 2
Número e identificación a nivel de género de ASVs detectados en caldos suplementados con 10ug/ml de oxitetraciclina cultivados a partir de muestras crudas del proceso de vermicompost analizados en el presente estudio. Costa Rica. 2023
|
Filo |
Género |
CM-TB |
CMC-TB |
VC-TB |
VCT-TB |
VCTM-TB |
|
Actinobacteria |
Bifidobacterium |
1 |
0 |
0 |
0 |
0 |
|
Bacteroidetes |
Myroides |
0 |
0 |
3 |
1 |
0 |
|
Bacteroidetes |
unc. Bacteroidaceae |
3 |
0 |
0 |
0 |
0 |
|
Bacteroidetes |
Chryseobacterium |
0 |
0 |
1 |
1 |
1 |
|
Bacteroidetes |
unc. Bacteroidales |
2 |
0 |
0 |
0 |
0 |
|
Bacteroidetes |
Prevotella |
0 |
2 |
0 |
0 |
0 |
|
Bacteroidetes |
unc. Saprospiraceae |
0 |
0 |
0 |
1 |
0 |
|
Bacteroidetes |
Bacteroides |
1 |
0 |
0 |
0 |
0 |
|
Bacteroidetes |
unc. Muribaculaceae |
1 |
0 |
0 |
0 |
0 |
|
Bacteroidetes |
Alloprevotella |
1 |
0 |
0 |
0 |
0 |
|
Bacteroidetes |
Faecalibacter |
1 |
0 |
0 |
0 |
0 |
|
Firmicutes |
Clostridium |
2 |
1 |
0 |
3 |
0 |
|
Firmicutes |
Kurthia |
2 |
0 |
0 |
1 |
0 |
|
Firmicutes |
Streptococcus |
2 |
0 |
0 |
0 |
0 |
|
Firmicutes |
Bacillus |
0 |
1 |
0 |
1 |
0 |
|
Firmicutes |
Solibacillus |
0 |
1 |
0 |
1 |
0 |
|
Firmicutes |
Rummeliibacillus |
0 |
1 |
0 |
0 |
0 |
|
Firmicutes |
Paeniclostridium |
1 |
0 |
0 |
0 |
0 |
|
Firmicutes |
Turicibacter |
1 |
0 |
0 |
0 |
0 |
|
Firmicutes |
Phascolarctobacterium |
1 |
0 |
0 |
0 |
0 |
|
Proteobacteria |
Aeromonas |
3 |
0 |
0 |
8 |
8 |
|
Proteobacteria |
Acinetobacter |
6 |
2 |
0 |
2 |
7 |
|
Proteobacteria |
Escherichia-Shigella |
1 |
1 |
0 |
1 |
1 |
|
Proteobacteria |
Proteus |
1 |
0 |
0 |
1 |
1 |
|
Proteobacteria |
Pseudomonas |
0 |
0 |
0 |
1 |
1 |
|
Proteobacteria |
Comamonas |
0 |
0 |
1 |
0 |
0 |
|
Proteobacteria |
Shewanella |
0 |
0 |
0 |
1 |
0 |
|
Proteobacteria |
Succinivibrio |
1 |
0 |
0 |
0 |
0 |
|
Proteobacteria |
Citrobacter |
1 |
0 |
0 |
0 |
0 |
|
Spirochaetes |
Treponema |
0 |
1 |
0 |
0 |
0 |
DISCUSIÓN
El uso de antibióticos en animales de granja como promotores del crecimiento o como medios inespecíficos de prevención y tratamiento de infecciones, ha impulsado el consumo de antibióticos y la resistencia entre las bacterias del hábitat animal (Economou & Gousia, 2015; Virto, et al., 2022).
En este estudio observamos crecimiento en caldo suplementado con oxitetraciclina, a partir de muestras del proceso de vermicompostaje de excretas de vaca y en los tés de vermicompost. Utilizamos 10µg/ml de oxitetraciclina para seleccionar bacterias resistentes ambientales. Esta concentración es superior a la concentración mínima inhibitoria (CMI) para bacterias susceptibles, pero menor a la CMI para bacterias resistentes patógenas humanas, considerando que los microorganismos de origen ambiental están expuestos a concentraciones mucho menores que bacterias de origen clínico y que también generan resistencia en el entorno natural (Popowska et al., 2012; Sandegren, 2014; Nogrado et al., 2021).
La muestra de excretas frescas fue la que presentó mayor cantidad de ASVs y géneros de bacterias resistentes, siendo la mayoría de estos taxones reportados en la microbiota normal de rumen y excretas, (Girija et al.,2013; Manyi-Loh et al., 2016). La presencia de bacterias resistentes y genes de resistencia, en excretas de ganado, incluyendo la resistencia a tetraciclina, se ha reportado anteriormente (Kobashi et al., 2007; Kyselková et al., 2015; Virto, et al., 2022), lo que coincide con los datos de este estudio. Este hallazgo podría relacionarse a que el ganado bovino de leche es sometido a tratamientos terapéuticos donde la tetraciclina es uno de los antibióticos aprobados (Carmona & Vindas, 2008). Por otro lado, a pesar de que los animales en tratamiento son separados, el manejo de las excretas de animales enfermos y no medicados es el mismo, pudiendo ambos llegar al proceso de vermicompostaje.
La presencia de coliformes como Escherichia en excretas no es de extrañar, ya que es parte de la microbiota normal de animales sanos y enfermos (Manyi-loh et al. 2016), sin embargo, se debe estudiar a mayor detalle, especialmente por su abundancia en esta muestra y por presentar un nivel de resistencia a oxitetraciclina. Cabe destacar que el proceso de precompostaje disminuyó en gran medida la abundancia de esta bacteria y no se detectaron secuencias de este ASV en la muestra de vermicompost. Otra bacteria abundante en CM-TB fue Kurthia massiliensis, bacteria Gram positiva normalmente encontrada en heces. Kurthia ha sido reportada anteriormente en cultivos suplementados con tetracilina, en heces de cerdos y pollos en granjas de producción (Kobashi et al., 2007), no obstante, no fue detectada en ninguna otra muestra del proceso.
La etapa del precompostaje se utiliza para disminuir posibles patógenos y sustancias tóxicas en las excretas que podrían afectar las lombrices (Mupondi et al. 2011). Durante este proceso de 15 días, se dan condiciones aerobias y termofílicas, que afectan a las bacterias anaerobias estrictas de la comunidad ruminal presentes en CM-TB, como Bacteroides, Bifidobacterium y Alloprevotella. Por otro lado, la temperatura puede reducir la población bacteriana de las excretas (Mupondi et al. 2011), incluyendo las bacterias resistentes. Esta disminución favorecería además la multiplicación de microorganismos colonizadores provenientes del ambiente (Wong et al., 2016), como lo son bacterias productoras de esporas abundantes en suelos y plantas como Bacillus, Solibacillus y Clostridium, encontradas en esta muestra.
En nuestro estudio, el vermicompost mostró la menor cantidad de géneros y ASVs resistentes de todas las muestras. La disminución de bacterias resistentes y genes de resistencia mediante el proceso de vermicompostaje ha sido reportada anteriormente (Cui et al., 2018; Huang et al., 2018; Wang et al., 2019). Esta atenuación se ha asociado con la disminución de la abundancia y la actividad bacteriana causada por la lombriz (Cui et al., 2018), con factores ambientales y cambios en la composición de la comunidad bacteriana de las excretas durante el vermicompostaje (Huang et al., 2018). No obstante, Myroides, el género de mayor abundancia en VC-TB, ha sido encontrado en intestinos de insectos, ambientes acuáticos y también se ha descrito en vermicompost (Dominguez et al. 2021). Las bacterias de este género son patógenos oportunistas poco frecuentes y, en China, se han reportado aislamientos resistentes a la mayoría de los antibióticos disponibles (Hu et al., 2016), por lo que es conveniente continuar estudiando su presencia en el vermicompost.
El proceso de preparación de los tés de vermicompost provocó el aumento de los ASVs resistentes a oxitetraciclina con respecto al VC-TB, además que favoreció el crecimiento de diversas especies de Acinetobacter y Aeromonas. Acinetobacter es un género ubicuo en la naturaleza encontrado también en excretas de vacas lecheras, compost, vermicompost, té de vermicompost e intestino de la lombriz Eisenia fetida (Fritz et al 2012; Girija et al. 2013; Pandey et al. 2018; Pérez-Pérez et al., 2018). Este género contiene especies oportunistas y patógenas como A. baumanni, y se han aislado cepas de esta especie resistentes a carbapenem a partir de vegetales (Adewoyin & Okoh, 2018), por lo que es importante analizar la presencia de ASVs clasificados como A. colistiniresistens presentes en alta abundancia en estas muestras y CM-TB
En el caso de Aeromonas, es un género de bacterias ubicuas, además con importancia como patógenos humanos, animales y alimentarios (Janda & Abbott, 2010; Grilo et al., 2020). Este género ha sido reportado en muestras provenientes del intestino de E. fetida (Toyota & Kimura, 2000; Furlong et al., 2002; Liu et al., 2011; Pérez-Pérez et al., 2018), de otras lombrices (Wust et al., 2011) y también en vermicompost (Blomström et al., 2016). Los ASVs identificados como Aeromonas detectados en caldo con oxitetraciclina, tienen similitud con accesiones procedentes de estos mismos ambientes, sin embargo, en el caso de los ASVs 2059, 2063 y 2057, también son similares a secuencias de Aeromonas aisladas a partir de un biofilm aeróbico sometido a estrés de oxitetraciclina (MK656380), además de otras secuencias de estudios de resistencia a antibióticos. Debido a la abundancia de este género en el té y en todas las muestras excepto el vermicompost, se podría especular que niveles subinhibitorios de tetraciclina, podrían estar presentes de manera constante en este sistema de producción lechero, lo que seleccionaría bacterias con capacidad de sobrevivir bajo estas concentraciones (Toyota & Kimura, 2000; Janda & Abbott, 2010; Grilo et al. 2020; Sandegren, 2014).
Nuestros resultados demuestran una comunidad bacteriana cultivable resistente a 10ug/ml de oxitetraciclina, presente en diferentes etapas del vermicompostaje de excretas bovinas, lo que puede indicar un proceso acumulativo de tratamientos con este antibiótico en los semovientes del sistema. A pesar de que se observó en las excretas frescas una mayor población resistente, las etapas de precompostaje y vermicompostaje influyeron en la disminución de los ASVs resistentes a 10ug/ml de oxitetraciclina, sin llegar a su eliminación. La presencia de Aeromonas, Escherichia-Shigella, Clostridium, Bacillus y Proteus en caldos suplementados con este antimicrobiano obtenidos a partir de los tés y excretas frescas, requiere mayor estudio con el fin de confirmar los resultados identificando los genes y determinantes de resistencia presentes en el sistema y así evaluar la funcionalidad e inocuidad de estos abonos.
AGRADECIMIENTOS
Agradecemos a todas las personas que apoyaron en diferentes etapas de la propuesta. Este trabajo fue financiado por Sistema de Estudios de Posgrado de la Universidad de Costa Rica.
ÉTICA, CONFLICTO DE INTERESES Y DECLARACIÓN DE FINANCIAMIENTO
Los autores declaran que han cumplido todos los requisitos éticos y legales pertinentes, tanto durante el estudio como en la elaboración del manuscrito; que no existen conflictos de intereses de ningún tipo; que todas las fuentes de financiación se indican completa y claramente en la sección de agradecimientos; y que están totalmente de acuerdo con la versión final editada del artículo.
La declaración de la contribución de cada autor al manuscrito es la siguiente: L.U-L. y L.F.A: Diseño del estudio. L.U-L. D.V-B, L.U. y R-W-J: Recogida de datos. L.U-L. Análisis de datos. Todos los coautores: preparación y aprobación final del manuscrito.
REFERENCIAS
Adewoyin, M. A., & Okoh, A. I. (2018). The natural environment as a reservoir of pathogenic and non-pathogenic Acinetobacter species. Reviews on Environmental Health, 33(3), 265-272. https://doi.org/10.1515/reveh-2017-0034
Aira, M., Olcina, J., Pérez-Losada, M., & Domínguez, J. (2016). Characterization of the bacterial communities of casts from Eisenia andrei fed with different substrates. Applied Soil Ecology, 98, 103-111. https://doi.org/10.1016/j.apsoil.2015.10.002
Arancon, N. Q., Edwards, C. A., Dick, R., & Dick, L. (2007). Vermicompost tea production and plant growth impacts. Biocycle 48, 51-52. https://bit.ly/3IaYcjR
Arancon, N. Q., Owens, J. D., & Converse, C. (2019). The effects of vermicompost tea on the growth and yield of lettuce and tomato in a non-circulating hydroponics system. Journal of plant nutrition, 42(19), 2447-2458. https://doi.org/10.1080/01904167.2019.1655049
Araya, M., Davidovich, G., Chaves, C., & Arias, M. L. (2005). Identificación de Enterococcus sp. en muestras de leche cruda del Área Metropolitana de Costa Rica y evaluación del patrón de sensibilidad a antibióticos. Archivos latinoamericanos de nutrición, 55(2), 161-166.
Artavia, S., Uribe, L., Saborío, F., Arauz, L. F., & Castro, L. (2010). Efecto de la aplicación de abonos orgánicos en la supresión de Pythium myriotylum en plantas de tiquisque (Xanthosoma sagittifolium). Agronomía Costarricense, 34(1), 17-29.
Aust, M. O., Godlinski, F., Travis, G. R., Hao, X., McAllister, T. A., Leinweber, P., & Thiele-Bruhn, S. (2008). Distribution of sulfamethazine, chlortetracycline and tylosin in manure and soil of Canadian feedlots after subtherapeutic use in cattle. Environmental pollution, 156(3), 1243-1251. https://doi.org/ 10.1016/j.envpol.2008.03.011
Black, Z., Balta, I., Black, L., Naughton, P. J., Dooley, J. S., & Corcionivoschi, N. (2021). The fate of foodborne pathogens in manure treated soil. Frontiers in Microbiology, 12, 781357. https://doi.org/10.3389/fmicb.2021.781357
Blomström, A. L., Lalander, C., Komakech, A. J., Vinnerås, B., & Boqvist, S. (2016). A metagenomic analysis displays the diverse microbial community of a vermicomposting system in Uganda. Infection ecology & epidemiology, 6(1), 32453. https://doi.org/10.3402/iee.v6.32453
Bolyen, E., Rideout, J. R., Dillon, M. R., Bokulich, N. A.,Abnet, C. C., Al-Ghalith, G. A., Alexander, H., Alm, E. J., Arumugam, M., Asnicar, F., Bai, Y., Bisanz, J. E., Bittinger, K., Brejnrod, A., Brislawn, C. J., Brown, C. T., Callahan, B. J., Caraballo-Rodríguez, A. M., Chase, J., Cope, E. K., Da Silva, R., … & Caporaso J. G. (2019). Reproducible, interactive, scalable and extensible microbiome data science using QIIME 2. Nature biotechnology, 37(8), 852-857. https://doi.org/10.1038/s41587-019-0209-9
Calderón, J. (1992) Determinación de residuos de sustancias inhibitorias en leche bovina no pausterizada. [Tesis de Licenciatura, Universidad Nacional de Costa Rica].
Callahan, B. J., McMurdie, P. J., & Holmes, S. P. (2017). Exact sequence variants should replace operational taxonomic units in marker-gene data analysis. The ISME journal, 11(12), 2639-2643. https://doi.org/10.1038/ismej.2017.119
Carmona, G., & Vindas, S. (2008). Uso racional de medicamentos veterinarios en ganado bovino. https://images.engormix.com/s_articles/carmonasolano_medicamentos.pdf
Casey, J. A., Curriero, F. C., Cosgrove, S. E., Nachman, K. E., & Schwartz, B. S. (2013). High-density livestock operations, crop field application of manure, and risk of community-associated methicillin-resistant Staphylococcus aureus infection in Pennsylvania. JAMA internal medicine, 173(21), 1980-1990. https://doi.org/10.1001/jamainternmed.2013.10408
Carrel, M., Schweizer, M. L., Sarrazin, M. V., Smith, T. C., & Perencevich, E. N. (2014). Residential proximity to large numbers of swine in feeding operations is associated with increased risk of methicillin-resistant Staphylococcus aureus colonization at time of hospital admission in rural Iowa veterans. Infection Control & Hospital Epidemiology, 35(2), 190-192. https://doi.org/10.1086/674860
Chun, J., Kim, K. Y., Lee, J. H., & Choi, Y. (2010). The analysis of oral microbial communities of wild-type and toll-like receptor 2-deficient mice using a 454 GS FLX Titanium pyrosequencer. BMC microbiology, 10(1), 1-8. https://doi.org/10.1186/1471-2180-10-101
Clarke, K. R., & Gorley, R. N. (2015). PRIMER v7: User Manual/Tutorial. https://bit.ly/44BoeWU
CLSI (2017). Performance Standards for Antimicrobial Susceptibility Testing; Twenty-Seven Informational Supplement. M100-S27. Wayne, PA: Clinical and Laboratory Standards Institute.
Cui, G., Li, F., Li, S., Bhat, S. A., Ishiguro, Y., Wei, Y., Yamada, T., Fu, X, & Huang, K. (2018). Changes of quinolone resistance genes and their relations with microbial profiles during vermicomposting of municipal excess sludge. Science of the total environment, 644, 494-502. https://doi.org/10.1016/j.scitotenv.2018.07.015
Domínguez, J., Aira, M., Crandall, K. A., & Pérez-Losada, M. (2021). Earthworms drastically change fungal and bacterial communities during vermicomposting of sewage sludge. Scientific Reports, 11(1), 15556. https://doi.org/10.1038/s41598-021-95099-z
Duffy, B., Sarreal, C., Ravva, S., & Stanker, L. (2004). Effect of molasses on regrowth of E. coli O157: H7 and Salmonella in compost teas. Compost science & utilization, 12(1), 93-96. https://doi.org/ 10.1080/1065657X.2004.10702163
Eastman, B. R., Kane, P. N., Edwards, C. A., Trytek, L., Gunadi, B., Stermer, A. L., & Mobley, J. R. (2001). The effectiveness of vermiculture in human pathogen reduction for USEPA biosolids stabilization. Compost Science & Utilization, 9(1), 38-49. https://doi.org/10.1080/1065657X.2001.10702015
Economou, V., & Gousia, P. (2015). Agriculture and food animals as a source of antimicrobial-resistant bacteria. Infection and drug resistance, 8, 49-62. https://doi.org/10.2147/IDR.S55778
Fritz, J. I., Franke-Whittle, I. H., Haindl, S., Insam, H., & Braun, R. (2012). Microbiological community analysis of vermicompost tea and its influence on the growth of vegetables and cereals. Canadian journal of microbiology, 58(7), 836-847. https://doi.org/10.1139/w2012-061
Furlong, M. A., Singleton, D. R., Coleman, D. C., & Whitman, W. B. (2002). Molecular and Culture-Based Analyses of Prokaryotic Communities from an Agricultural Soil and the Burrows and Casts of the Earthworm Lumbricus rubellus. Applied and Environmental Microbiology, 68(3), 1265-1279. https://doi.org/10.1128/AEM.68.3.1265-1279.2002
Giacomini, D. G. (2012). Formal recommendation by the National organic Standard Board (NOSB) to the National Organic Program (NOP). US Department of Agriculture.
Girija, D., Deepa, K., Xavier, F., Antony, I., & Shidhi, P. R. (2013). Analysis of cow dung microbiota—a metagenomic approach. Indian Journal of Biotechnology, 12, 372-378. https://bit.ly/44AC7of
Grilo, M. L., Sousa-Santos, C., Robalo, J., & Oliveira, M. (2020). The potential of Aeromonas spp. from wildlife as antimicrobial resistance indicators in aquatic environments. Ecological Indicators, 115, 106396. https://doi.org/10.1016/j.ecolind.2020.106396
Gupta, K. K., Aneja, K. R., & Rana, D. (2016). Current status of cow dung as a bioresource for sustainable development. Bioresources and Bioprocessing, 3, 1-11. https://doi.org/10.1186/s40643-016-0105-9
Gutiérrez, K., Alfaro, M., Granados, F., Sánchez, J., García, F., & Rodríguez, C. (2010). Detección de tetraciclinas en nueve lotes de alimentos para cerdos, tilapias y pollos producidos en Costa Rica: incumplimiento de normativas y disconformidades con el etiquetado oficial de garantía. Agronomía Costarricense, 34(2), 145-151
Hu, S. H., Yuan, S. X., Qu, H., Jiang, T., Zhou, Y. J., Wang, M. X., & Ming, D. S. (2016). Antibiotic resistance mechanisms of Myroides sp. Journal of Zhejiang University-Science B, 17(3), 188-199. https://doi.org/ 10.1631/jzus.B1500068
Hu, J., Zhao, H., Wang, Y., Yin, Z., & Kang, Y. (2020). The bacterial community structures in response to the gut passage of earthworm (Eisenia fetida) feeding on cow dung and domestic sludge: Illumina high-throughput sequencing-based data analysis. Ecotoxicology and Environmental Safety, 190, 110149. https://doi.org/10.1016/j.ecoenv.2019.110149
Huang, K., Xia, H., Wu, Y., Chen, J., Cui, G., Li, F., Chen, Y., & Wu, N. (2018). Effects of earthworms on the fate of tetracycline and fluoroquinolone resistance genes of sewage sludge during vermicomposting. Bioresource technology, 259, 32-39. https://doi.org/10.1016/j.biortech.2018.03.021
Huijsdens, X. W., Van Dijke, B. J., Spalburg, E., van Santen-Verheuvel, M. G., Heck, M. E., Pluister, G. N., Voss, A., Wannet, W., & De Neeling, A. J. (2006). Community-acquired MRSA and pig-farming. Annals of clinical microbiology and antimicrobials, 5, 1-4. https://doi.org/10.1186/1476-0711-5-26
Janda, J. M., & Abbott, S. L., (2010). The genus Aeromonas: taxonomy, pathogenicity, and infection. Clinical Microbiology Reviews, 23(1), 35-73. https://doi.org/10.1128/CMR.00039-09
Kiyasudeen, K., Ibrahim, M. H., Quaik, S., Ismail, S. A. (2016). Vermicompost, its applications and derivatives. In K. Kiyasudeen, M. Ibrahim, S. Quaik, S. Ismail (Eds.), Prospects of Organic Waste Management and the Significance of Earthworms (pp. 201-230). Springer.
Kraemer, S. A., Ramachandran, A., & Perron, G. G. (2019). Antibiotic pollution in the environment: from microbial ecology to public policy. Microorganisms, 7(6), 180. https://doi.org/10.3390/microorganisms7060180
Kumar, K., Gupta, S. C., Chander, Y., & Singh, A. K. (2005). Antibiotic use in agriculture and its impact on the terrestrial environment. Advances in Agronomy, 87, 1-53. https://doi.org/10.1016/S0065-2113(05)87001-4
Kobashi, Y., Hasebe, A., Nishio, M., & Uchiyama, H. (2007). Diversity of tetracycline resistance genes in bacteria isolated from various agricultural environments. Microbes and environments, 22(1), 44-51.
Kyselková, M., Jirout, J., Vrchotová, N., Schmitt, H., & Elhottová, D. (2015). Spread of tetracycline resistance genes at a conventional dairy farm. Frontiers in microbiology, 6, 536. https://doi.org/10.3389/fmicb.2015.00536
Li, Z., Chen, C., Zhang, K., Zhang, Z., Zhao, R., Han, B., Yang, F., & Ding, Y. (2022). Response of Antibiotic Resistance Genes and Related Microorganisms to Arsenic during Vermicomposting of Cow Dung. International Journal of Environmental Research and Public Health, 19(21), 14475. https://doi.org/10.3390/ijerph192114475
Liu, D., Lian, B., Wang, B., & Jiang, G. (2011). Degradation of potassium rock by earthworms and responses of bacterial communities in its gut and surrounding substrates after being fed with mineral. PLoS One, 6(12), e28803. https://doi.org/10.1371/journal.pone.0028803
Manyi-Loh, C. E., Mamphweli, S. N., Meyer, E. L., Makaka, G., Simon, M., & Okoh, A. I. (2016). An overview of the control of bacterial pathogens in cattle manure. International journal of environmental research and public health, 13(9), 843. https://doi.org/10.3390/ijerph13090843
Mupondi, L. T., Mnkeni, P. N., & Muchaonyerwa, P. (2011). Effects of a precomposting step on the vermicomposting of dairy manure-waste paper mixtures. Waste Management & Research, 29(2), 219-228. https://doi.org/10.1177/0734242X10363142
Nogrado, K., Unno, T., Hur, H. G., & Lee, J. H. (2021). Tetracycline-resistant bacteria and ribosomal protection protein genes in soils from selected agricultural fields and livestock farms. Applied Biological Chemistry, 64(1), 1-9. https://doi.org/10.1186/s13765-021-00613-6
Organización de las Naciones Unidad para la Alimentación y la Agricultura (FAO). (2021). El Plan de acción de la FAO sobre la resistencia a los antimicrobianos 2021-2025. https://doi.org/10.4060/cb5545es
Oliver, J. P., Gooch, C. A., Lansing, S., Schueler, J., Hurst, J. J., Sassoubre, L., Crossette, E. M., & Aga, D. S. (2020). Invited review: Fate of antibiotic residues, antibiotic-resistant bacteria, and antibiotic resistance genes in US dairy manure management systems. Journal of dairy science, 103(2), 1051-1071. https://doi.org/10.3168/jds.2019-16778
Pandey, P., Chiu, C., Miao, M., Wang, Y., Settles, M., Silva del Rio, N., Castillo, A., Souza, A., Pereira, R., & Jeannotte, R. (2018). 16S rRNA analysis of diversity of manure microbial community in dairy farm environment. PloS one, 13(1), e0190126. https://doi.org/10.1371/journal.pone.0190126
Pane, C., Celano, G., & Zaccardelli, M. (2014). Metabolic patterns of bacterial communities in aerobic compost teas associated with potential biocontrol of soilborne plant diseases. Phytopathologia Mediterranea, 53(2), 277-286. https://doi.org/10.14601 / Phytopathol_Mediterr-13363
Pant, A. P., Radovich, T. J., Hue, N. V., & Paull, R. E. (2012). Biochemical properties of compost tea associated with compost quality and effects on pak choi growth. Scientia horticulturae, 148, 138-146. https://doi.org/10.1016/j.scienta.2012.09.019
Pathma, J., & Sakthivel, N. (2012). Microbial diversity of vermicompost bacteria that exhibit useful agricultural traits and waste management potential. SpringerPlus, 1, 1-19. https://doi.org/10.1186/2193-1801-1-26
Pérez-Pérez, J. A., Espinosa-Victoria, D., Silva-Rojas, H. V., & López-Reyes, L. (2018.) Diversidad de la microbiota bacteriana cultivable del tracto digestivo de Eisenia fetida. Revista fitotecnia mexicana, 41(3), 255-264. https://doi.org/10.35196/rfm.2018.3.255-264
Popowska, M., Rzeczycka, M., Miernik, A., Krawczyk-Balska, A., Walsh, F., & Duffy, B. (2012). Influence of soil use on prevalence of tetracycline, streptomycin, and erythromycin resistance and associated resistance genes. Antimicrobial agents and chemotherapy, 56(3), 1434-1443. https://doi.org/ 10.1128/AAC.05766-11
Quast, C., Pruesse, E., Yilmaz, P., Gerken, J., Schweer, T., Yarza, P., Peplies, J., & Glöckner, F. O. (2012). The SILVA ribosomal RNA gene database project: improved data processing and web-based tools. Nucleic acids research, 41(D1), D590-D596. https://doi.org/10.1093/nar/gks1219
Romero-Tepal, E. M., Contreras-Blancas, E., Navarro-Noya, Y. E., Ruíz-Valdiviezo, V. M., Luna-Guido, M., Gutiérrez-Miceli, F. A., & Dendooven, L. (2014). Changes in the bacterial community structure in stored wormbed leachate. Microbial Physiology, 24(2), 105-113 https://doi.org/ 10.1159/000357915
Sandegren, L. (2014). Selection of antibiotic resistance at very low antibiotic concentrations. Upsala journal of medical sciences, 119(2), 103-107. https://doi.org/ 10.3109/03009734.2014.904457
Scheuerell, S., & Mahaffee, W. (2002). Compost tea: principles and prospects for plant disease control. Compost Science & Utilization, 10(4), 313-338.
Scheuerell, S. J., & Mahaffee, W. F. (2004). Compost tea as a container medium drench for suppressing seedling damping-off caused by Pythium ultimum. Phytopathology, 94(11), 1156-1163.
Silbergeld, E. K., Graham, J., & Price, L. B. (2008). Industrial food animal production, antimicrobial resistance, and human health. Annu. Rev. Public Health, 29, 151-169. https://doi.org/10.1146/annurev.publhealth.29.020907.090904
Tortós, C. L., Correa, M. T., & Guerra, H. C. (2006). Estudio sobre el manejo de antibióticos en hatos lecheros artesanales de Costa Rica. Ciencias Veterinarias, 24(1), 91-106.
Toyota, K., & Kimura, M. (2000). Microbial community indigenous to the earthworm Eisenia foetida. Biology and fertility of soils, 31(3), 187-190.
Uribe, L., Arauz, L. F., Mata, M., Meneses, G., & Castro, L. (2009). Efecto del vermicompostaje sobre las poblaciones de Colletotrichum acutatum y Pectobacterium carotovorum presentes en residuos de plantas. Agronomía Costarricense, 33(1), 91-101. https://www.redalyc.org/pdf/436/43612054008.pdf
Vindas, M. J. (2019). Espectro de resistencia, estructura genética y relaciones evolutivas de plásmidos conjugativos con resistencia a tetraciclinas aislados de ecosistemas agropecuarios costarricenses. [Tesis Maestría Académica en Microbiología]. Universidad de Costa Rica.
Virto, M., Santamarina-García, G., Amores, G., & Hernández, I. (2022). Antibiotics in Dairy Production: Where Is the Problem? Dairy, 3(3), 541-564. https://doi.org/10.3390/dairy3030039
Wang, Y., Yin, Z., Zhao, H., Hu, J., & Kang, Y. (2019). The effects of tetracycline concentrations on tetracycline resistance genes and their bacterial hosts in the gut passages of earthworms (Eisenia fetida) feeding on domestic sludge. Environmental Science and Pollution Research, 26, 34412-34420. https://doi.org/10.1007/s11356-019-06495-y
Wong, K., Shaw, T. I., Oladeinde, A., Glenn, T. C., Oakley, B., & Molina, M. (2016). Rapid microbiome changes in freshly deposited cow feces under field conditions. Frontiers in microbiology, 7, 500. https://doi.org/10.3389/fmicb.2016.00500
Wüst, P. K., Horn, M. A., & Drake, H. L. (2011). Clostridiaceae and Enterobacteriaceae as active fermenters in earthworm gut content. The ISME journal, 5(1), 92-106. https://doi.org/10.1038/ismej.2010.99
Yatoo, A. M., Ali, M., Baba, Z. A., & Hassan, B. (2021). Sustainable management of diseases and pests in crops by vermicompost and vermicompost tea. A review. Agronomy for Sustainable Development, 41(1), 1-26. https://doi.org/10.1007/s13593-020-00657-w
Yoon, S. H., Ha, S. M., Kwon, S., Lim, J., Kim, Y., Seo, H., & Chun, J. (2017). Introducing EzBioCloud: a taxonomically united database of 16S rRNA gene sequences and whole-genome assemblies. International journal of systematic and evolutionary microbiology, 67(5), 1613. https://doi.org/10.1099/ijsem.0.001755
Zalewska, M., Błażejewska, A., Czapko, A., & Popowska, M. (2021). Antibiotics and antibiotic resistance genes in animal manure–consequences of its application in agriculture. Frontiers in Microbiology, 12, 610656. https://doi.org/10.3389/fmicb.2021.610656
Zamora, K., Castro, L., Wang, A., Arauz, L. F., & Uribe, L. (2017). Uso potencial de lixiviados y tés de vermicompost en el control del ojo de gallo del cafeto Mycena citricolor. Agronomía Costarricense, 41(1), 33-51.