
Identificación y caracterización molecular de cianobacterias tropicales
de los géneros Nostoc, Calothrix, Tolypothrix y Scytonema
(Nostocales: Nostocaceae), con posible potencial biotecnológico
Mariela Corrales Morales1, Karolina Villalobos1, Abad Rodríguez Rodríguez 2, Nelson Muñoz Simón3
& Rodolfo Umaña-Castro1
1. Laboratorio de Análisis Genómico, Escuela de Ciencias Biológicas, Universidad Nacional, Sede Omar Dengo, Heredia, Costa Rica;
mari.corrales19@gmail.com, karo26vc@gmail.com, rodolfo.umana.castro@una.cr
2. Laboratorio de Biotecnología de Microalgas, Escuela de Ciencias Biológicas, Universidad Nacional, Sede Omar Dengo, Heredia, Costa Rica; abadrr@gmail.com
3. Escuela de Ciencias Biológicas, Universidad Nacional, Sede Omar Dengo, Heredia, Costa Rica; nelsonmuns@hotmail.com
Recibido 23-iii-2017 • Corregido 19-vii-2017 • Aceptado 21-vii-2017
ABSTRACT: Identification and molecular characterization of tropical cyanobacteria of the genus Nostoc, Calothrix, Tolypothrix and Scytonema (Nostocales: Nostocaceae), with possible biotechnological potential. Cyanobacteria or “Blue-Green” algae comprise a diverse cluster of prokaryotic photoautotrophs that share a high morphological and molecular abundance. Biotechnological advances on nitrogen fixation, bioremediation, pharmaceutical and nutritional value have been developed. Traditional identification based on morphology has been the most common applied technique, but it may be inaccurate because of the phenotypic plasticity that those organisms present. For this reason, molecular techniques had become robust tools for taxonomic positioning of tropical cyanobacterial isolates, associated with morphology identification. This study focuses on the morphological identification and molecular characterization of cyanobacteria isolated in different tropical environments of Costa Rica, for biotechnological prospects. Strains were grown in BG0-11 media, photographed under light microscope and classified at genera level. Molecular identification was carried by PCR and sequencing using DNA-dependent RNA polymerase (rpoC1) and small-subunit ribosomal RNA (16S rRNA) gen primers. Subsequently, a phylogenetic positioning analysis was performed by MAFFTv.7 alignment and maximum likelihood by raxMLGUI. Based on phenotypic characteristics, four genera of Nostocales were identified: Calothrix sp., Tolypothrix sp., Scytonema sp. and Nostoc sp. Molecular analysis shows a taxonomic grouping that is consistent with the observed morphology, with the 16S rDNA gene yielding robust and stable results. The identified genera are known for their nitrogen fixation capability: Nostoc and Calothrix have been used in maintaining fertility and soil recovering studies. Nostoc is known for its nutritional properties and biofuel production; a characteristic shared by Tolypothrix as well. The last genera; Scytonema, it is known for its pharmacological properties. With the successful identification of cyanobacteria strains isolated from tropical environments, it is possible to continue the study of genes responsible for properties of biotechnological interest and to evaluate their potential, with future perspectives of application and biodiversity conservation.
Key words: Cyanobacteria, 16S rRNA, rpoC1, characterization molecular, morphological identification, biotechnological potential.
RESUMEN: Las cianobacterias son procariotas fotoautótrofos, morfológica y molecularmente diversas, en las cuales se han reportado avances biotecnológicos sobre fijación de nitrógeno, biorremediación y valor nutricional-farmacéutico. La identificación tradicional de cianobacterias basada en morfología puede ser imprecisa, por lo que las técnicas moleculares son una herramienta robusta para el posicionamiento taxonómico de estas cepas tropicales. Este estudio se enfoca en la identificación morfológica y caracterización molecular de cianobacterias de ambientes tropicales de Costa Rica, para perspectivas biotecnológicas futuras. Las cepas fueron cultivadas en medio BG0-11, fotografiadas en microscopio de luz y clasificadas a nivel de género. La identificación molecular se llevó a cabo utilizando cebadores del gen ARN polimerasa ADN-dependiente (rpoC1) y la subunidad pequeña ARN ribosomal (16S ARNr). Seguidamente, se realizó un análisis de posicionamiento filogenético mediante alineamiento múltiple con MAFFTv.7 y máxima-verosimilitud con raxMLGUI. Basados en características fenotípicas, se identificaron cuatro géneros de cianobacterias Nostocales: Calothrix sp., Tolypothrix sp., Scytonema sp. y Nostoc sp. Los géneros identificados son conocidos por su capacidad de fijación de nitrógeno y potencial nutricional, farmacológico e industrial. Con una identificación exitosa de cianobacterias de ambientes tropicales, es posible continuar con más estudios de sus propiedades biotecnológicas y evaluar su potencial, con perspectivas futuras de aplicación y conservación de la biodiversidad.
Palabras clave: Cianobacterias, 16S ARNr, rpoC1, caracterización molecular, identificación morfológica, potencial biotecnológico.
Las cianobacterias, anteriormente conocidas como algas verde azuladas, son un grupo heterogéneo de procariotas fotoautótrofos, las cuales realizan fotosíntesis oxigénica similar a las algas eucariotas y plantas (Singh, Pathak & Sinha, 2017). Estas ocupan hábitats diversos, como terrestres, aguas dulces y marinos, incluyendo medios extremófilos (Prasanna, Sood, Ratha, & Singh, 2014). Según Sharma (2015), los suelos de las zonas tropicales poseen gran diversidad de especies de cianobacterias, seguido de algas verde-amarillas y diatomeas. Estos microorganismos poseen diversas morfologías desde unicelulares a filamentosas hasta coloniales (Faldu et al., 2014). Dentro de la familia Nostocaceae se han descrito cinco géneros diazótrofos, entre ellos: Nostoc, Calothrix, Tolypothrix y Scytonema (Lee et al., 2014).
Se reportan estudios de caracterización molecular y morfológica de cianobacterias del orden Nostocales en suelos minerales y en regiones como Australia, el continente asiático, europeo y Sur América (Galhano et al., 2011; Arima et al., 2012; Quesada & Vincent, 2012; Silva & de Jesús, 2013).
Especies del género Nostoc están distribuidas desde los trópicos hasta las zonas polares y presentan simbiosis terrestre adaptándose a rocas, paredes de piedra y plantas (Teneva, Stoyanov, Mladenov & Dzhambazov, 2012; Liao, Wu, Tai, Chi & Lin, 2015). Estos microorganismos son capaces de sobrevivir a suelos pobres en nutrientes y condiciones de deshidratación debido a que poseen polisacáridos en su pared celular que los protegen contra dichas condiciones (Jensen et al., 2013).
Las cianobacterias del género Calothrix se distribuyen en regiones templadas, tropicales y subtropicales, desde hábitats marinos hasta dulceacuícolas. Además se han encontrado en afluentes y aguas estancadas, sujetos a rocas sumergidas o en la superficie o interior de troncos (Rinkel & Manoylov, 2014). Dentro del género Scytonema, sus especies son consideradas cosmopolitas, creciendo en hábitats tropicales y subtropicales. Un ejemplo de ello son los suelos lateríticos, rocas y reservorios de vegetación de agua (Sant’Anna et al., 2011; Komarek et al., 2014). Especies del género Tolipothrix se distribuyen en hábitats hipolíticos y endolíticos, como en campos agrícolas de Sur América (Quesada & Vincent, 2012; Silva & de Jesús, 2013).
Debido a la importancia en las posibles aplicaciones de las cianobacterias como recurso biológico, es relevante que, seguido del aislamiento y mantenimiento de las cepas, se realice la identificación taxonómica y la caracterización de los aislados (Galhano et al., 2011). Tradicionalmente se ha realizado una identificación basada en morfología; sin embargo, la misma puede ser imprecisa por la plasticidad fenotípica que estos microorganismos presentan (Lee et al., 2014); por ello, se han venido implementando técnicas moleculares para el posicionamiento taxonómico, en conjunto con las características fenotípicas. Para análisis de identificación y caracterización molecular, se reporta el uso de los genes que codifican para la subunidad gamma de la ARN polimerasa dependiente de ADN (rpoC), que contiene un componente central único que está ausente en las demás eubacterias (Lee et al., 2014). Por otro lado, se conoce el uso de la subunidad pequeña del ARN ribosomal (16S ARNr) para la construcción de filogenias de cianobacterias (Zhen, Liu, Rensing, Yan & Zhang, 2017).
En la actualidad, se han desarrollado avances biotecnológicos que han demostrado el potencial que poseen las cianobacterias en diversas áreas, convirtiéndose así en un grupo de alto valor económico (Singh et al., 2017). Entre estas aplicaciones se encuentra la capacidad de fijación de nitrógeno; este proceso se puede llevar a cabo en células especializadas y diferenciadas morfológicamente llamadas heterocistos (Flores, López-Lozano & Herrero, 2015). Dicha característica posee un alto valor agronómico utilizándose como biofertilizantes en campos de cultivo. De (1939), reportó el potencial de fijación de nitrógeno de ciertos géneros de cianobacterias, proponiendo un modelo de fertilidad natural del suelo en campos de arroz y su mantenimiento en el proceso de fijación biológica de nitrógeno por dichos microorganismos.
Por otra parte, se ha demostrado el potencial bioremediador asociado con la capacidad de degradar materiales o sustancias que se consideran contaminantes (Sharma, 2015). Una alternativa para el tratamiento de residuos ha sido el uso de la biomasa de las cianobacterias, ya que la pared celular de dichos microorganismos posee diversos grupos funcionales que les confiere carga negativa a la superficie celular, adsorbiendo así los iones metálicos que, en medio acuoso, se presentan en formas catiónicas (Gupta & Chaudhuri, 2016).
Este estudio se enfoca en la identificación morfológica y caracterización molecular de cianobacterias aisladas en diferentes ambientes tropicales de Costa Rica, para su evaluación a futuro como cepas promisorias en aplicaciones biotecnológicas.
MATERIALES Y MÉTODOS
Material Biológico: Se analizaron siete cepas de cianobacterias, aisladas de diferentes ambientes terrestres (Cuadro 1). La selección para su posterior caracterización morfológica y molecular se llevó acabo tomando en cuenta aquellos géneros en los que se presenta cierta ambigüedad taxonómica reportada para especies tropicales. Las cepas fueron cultivadas en medio líquido y agar BG0-11, durante 15 días, esto con el fin de evaluar cualitativamente una actividad biológica específica correspondiente a la fijación de nitrógeno, lo que se visualiza como la capacidad de generar un aumento de biomasa. Las cepas se conservan en el Laboratorio de Biotecnología de Microalgas de la Escuela de Ciencias Biológicas, las cuales se repican mensual o bimensualmente en tubos de agar BG11 inclinado. Cada cultivo fue observado y fotografiado bajo un microscopio óptico Nikon eclipse Ni y las cianobacterias presentes se clasificaron a nivel de género adoptando el sistema propuesto por Desikachary (1959) y Komárek y Anagnostidis (1989, 1999, 2005).
CUADRO 1
Sitio y año de aislamiento de las cianobacterias estudiadas.
|
Código* |
Sitio de aislamiento |
Año de aislamiento |
|
BGN04 |
Suelo arcilloso cafetalero, Barreal, Heredia |
1981 |
|
BGN17 |
Río Jorco, Desamparados, San José |
1982 |
|
BGN18 |
Estanque artificial de piscicultura, Cañas, Guanacaste |
1985 |
|
BGN21 |
Suelo cafetalero, Barreal, Heredia |
1983 |
|
BGN22 |
Bioincrustaciones (biofouling), Embalse Arenal, Alajuela |
1986 |
|
BGN23 |
Laguna Bosque Sánchez |
1994 |
|
BGN25 |
Suelo bosque, Reserva Biológica La Selva, Sarapiquí, Heredia |
1987 |
*Código de accesión en la Algoteca del Laboratorio de Biotecnología de Microalgas de la Universidad Nacional.
Extracción de ADN: El ADN genómico de las cepas de cianobacterias se aisló utilizando el protocolo de extracción por CTAB de Porebski, Bailey & Baum (1997), con ciertas modificaciones. Se tomó de 0,1-0,2g de biomasa, a la cual se adicionó 100µL de TE (10mM Tris, 1mM EDTA) y 3µL de proteinasa K (20mg/mL). Al contenido de cada tubo se le realizó una disrupción mecánica con balines de vidrio y metal, en un desmembrador Retsch mm400, a una agitación de 30Hz por 1min. El material homogenizado fue incubado a 37°C por una hora, seguidamente se adicionó 500µL de buffer CTAB 2X y se incubó a 65°C por 30min. Al lisado celular se le adicionó 500µL de Cloroformo-alcohol isoamílico (24:1) y se separaron las fases. La fase acuosa se recuperó y se agregó 0,5 volúmenes de NaCl 5M y 0,6 volúmenes de isopropanol para precipitar el ADN, seguido de una incubación a -20°C por una hora. El ADN recuperado se lavó con etanol 70%, se resuspendió en 100µL de agua libre de nucleasas (Ambion) y finalmente se realizó un tratamiento de eliminación de ARN mediante ARNasa Invitrogen (10µg/mL), según las instrucciones de manufactura. El ADN obtenido se cuantificó con un NanoDrop 2000 UV-Vis Thermo Scientific, se movilizó en un gel de agarosa al 1% (TBE 1X), 80V por una hora y se visualizó en un transiluminador UV Fisher Scientific.
Amplificación por PCR de los genes 16S ARNr y rpoC1: Las reacciones PCR se realizaron en un volumen final de 20µL, el cual contenía buffer GoTaq Flexi 1X, MgCl2 2,5mM, dNTPs 0,2mM de cada uno, 1U de GoTaq Flexi ADN polimerasa y 150ng de ADN genómico aproximadamente. Además de 0,5mM de cada uno de los cebadores sentido y antisentido. Los cebadores empleados para amplificar la región de la subunidad pequeña del ARN ribosomal (16S ARNr) fueron los CYAN106F (5’-CGGACGGGTGAGTAACGCGTGA-3’) y CYAN781R (5’-GACTACWGGGGTATCTAATCCCWTT-3’), con un producto esperado de 805pb (Zhen et al., 2017). Para la reacción de amplificación de la región de la subunidad gamma de la ARN polimerasa dependiente de ADN (rpoC), se emplearon los cebadores rpoC145F (5’-ACTCTGAARCCAGAAATGGA-3’) y rpoC1006R (5’-TGCTTACCTTCAATAATGTC-3’) con un producto esperado de 880pb (Lee et al., 2014). Las amplificaciones se realizaron en un termociclador Proflex Applied Biosystems, con un perfil térmico de 95°C por 5min, 35 ciclos de 94°C por 1min, 60°C-55°C por 45s (16S ARNr y rpoC1, respectivamente), 72°C por 1min, y una extensión final de 72°C por 5min. Los productos derivados de la PCR se movilizaron en un gel de agarosa al 1,5% (TBE 1X) y se visualizaron en un transiluminador UV Fisher Scientific.
Secuenciación de los amplicones y análisis filogenético: Se utilizó un protocolo alternativo de limpieza y purificación de productos de PCR mediante precipitación con etanol 95% y acetato de sodio 3M (Cuevas, Goyenechea & Iturbe, 2007). Los productos de PCR purificados fueron secuenciados en ambas direcciones en un analizador genético multicapilar 3130, BigDye Terminator™ V3.1, Applied Biosystems, en el Laboratorio de Análisis Genómico (LAGEN) de la Escuela de Ciencias Biológicas de la UNA. Las secuencias obtenidas fueron editadas con el programa bioinformático Geneious R9, Biomatters Ltd, Nueva Zelanda y analizadas utilizando la herramienta básica de búsqueda local de similitud (BLASTn, por sus siglas en inglés) (Altschul, Gish, Miller, Myers & Lipman, 1990) de la base de datos NCBI (http://www.ncbi.nlm.nih.gov/blast) mediante parámetros por defecto. Las secuencias obtenidas fueron depositadas en el GenBank (www.ncbi.nlm.nih.gov/genbank) con los siguientes números de accesión: MF581033 a MF581038.
Se realizó un análisis de posicionamiento filogenético entre las secuencias parciales de este estudio y las obtenidas en la base de datos Genbank. El árbol filogenético de las regiones 16S ARNr y rpoC1 se fabricaron derivados de un alineamiento con el programa en línea MAFFT 7.0 (http://mafft.cbrc.jp/alignment/server), con el método iterativo de refinamiento (FFT-NS-i) y el parámetro 1PAM / k = 2. Por otro lado, se utilizó el programa Gblocks (Castresana, 2002) para seleccionar bloques conservados (sin alineamientos erróneos, ni regiones divergentes) para el posterior análisis filogenético. Para determinar el mejor modelo de sustitución nucleotídica, el alineamiento se analizó con jModelTest 2.1.10 (Darriba, Taboada, Doallo & Posada, 2012) con la estrategia de selección mediante el criterio de información de Akaike (AIC). Posteriormente, se elaboró un árbol de posicionamiento filogenético utilizando el algoritmo de máxima verosimilitud (ML) mediante el programa raxmlGUI v.7.4.2 (Stamatakis, Ludwig & Meier, 2005) con el modelo reversible en tiempo general (GTR-GAMMA), empleando 2 000 repeticiones. Los árboles fueron visualizados y editados con el programa FigTree 1.4 (Rambaut, 2009).
RESULTADOS
Basados en características fenotípicas, de las siete cepas estudiadas, se identificaron cuatro géneros de cianobacterias Nostocales: Calothrix sp. presenta heterocistos basales, tricoma estrechado hacia la parte terminal, células terminales piliformes, difícilmente observables en cultivos, vaina relativamente delgada, con evidencia de laminación, múltiples hormogonios presentes en los cultivos. Dimensiones: 5µ x 5.8µ (Fig. 1A, 1B). Tolypothrix sp con filamentos heteropolares, ramificaciones falsas en Y y T, heterocistos mayormente terminales con un solo poro. Las dimensiones que se observan son de células vegetativas 4,7µ x 5µ (Fig. 1C). Scytonema sp. presenta filamentos con ramificación falsa y enrollados, vaina gruesa y lamelada, necridios solitarios o agrupados-continuos (Fig. 1D). Nostoc sp. la cepa BGN04 presenta heterocistos principalmente terminales, algunos intercalares, los terminales se acortan levemente hacia los extremos, su reproducción implica la formación de hormogonios de morfología muy diferente, constreñidos en las paredes y muy móviles. Las dimensiones observadas son de acinetos 6,6µ x 4,7µ, heterocistos 4,2µ x 3,7µ y células vegetativas 2,7µ x 3,2µ (Fig. 1E). La cepa BGN25 se diferencia por la presencia de tricomas muy pequeños envueltos por una cubierta mucilaginosa muy fina (Fig. 1F).
El análisis de posicionamiento filogenético entre las secuencias parciales de la subunidad pequeña del ARN ribosomal (16S ARNr) obtenidas y las derivadas del Genbank, evidencian un agrupamiento taxonómico que concuerda con la morfología observada en la Fig. 1. Por otro lado, el análisis de taxonomía molecular derivado de la región de la subunidad gamma de la ARN polimerasa dependiente de ADN (rpoC) mostró agrupamientos erráticos y poco soportados (dato no mostrado).
En la figura 2 se observan dos grandes clados que dividen la topología con soportes de las ramas de un 46% y 70%. El clado I que contiene a los géneros Scytonema y Calothrix; por otro lado el clado II que incluye a los géneros Anabaena, Nostoc, Tolypothrix y Trichormus. En el clado I (posicionado en el grupo superior de la topología), el aislado BGN23 está bien soportado junto con el agrupamiento de las especies del género Scytonema, específicamente a Scytonema geitleri. De igual manera las cepas BGN17, BGN18, BGN22 se asocian al nodo que derivan al género Calothrix sp., mostrándose una diferencia en el agrupamiento por especie entre BGN18 (asociado con Calothrix parietina, valor de soporte de la rama de un 89%) y BGN17-BGN22 (agrupadas con Calothrix brevissima, valor de soporte de la rama de un 87%).
En el clado II (localizado en la agrupación de nodos inferiores a la topología), se observa que las cepas BGN04 y BGN25 se asocian al agrupamiento de Nostoc sp., sin embargo, los dos aislados se posicionan en la especie Nostoc entophytum. Por otro lado, el aislado BGN21 se localiza en el grupo de Tolypothrix sp., vinculado con la especie T. tenuis. Finalmente, de las agrupaciones que corresponde a Anabaena sp. y Trichormus sp. no se observaron asociaciones taxonómicas directas con las cepas de este estudio.
DISCUSIÓN
Las cepas identificadas mediante la observación de características morfológicas se localizan entre las cianobacterias pertenecientes al orden Nostocales (Komarek et al., 2014). Hasta el momento, existen algunos hallazgos científicos que tratan sobre la taxonomía y la filogenia de algunos géneros particulares pertenecientes al orden abordado en nuestro estudio, dichos estudios demuestran que la clasificación tradicional debe ser revisada (Rajaniemi et al. 2005). Sihvonen et al. (2007) sugieren que Tolypothrix y Calothrix corresponden a géneros polifiléticos, mientras que Lyra et al. (2001), Gugger et al. (2002), Iteman, Rippka, de Marsac y Herdman (2002) y Rajaniemi et al. (2005) lo hacen para los géneros Anabaena, Trichormus y Aphanizomenon, por ello el uso de herramientas moleculares es de suma importancia para esclarecer las relaciones entre los diferentes géneros, en especial aquellos provenientes de las zonas tropicales (Komarek, com. pers).
En combinación con el método morfológico, el método de taxonomía molecular basado en la región ribosomal 16S generó suficiente información para determinar que las cepas BGN corresponden taxonómicamente con lo descrito en la identificación fenotípica de este estudio. Existen amplios reportes sobre el empleo del gen 16S ribosomal como herramienta para desarrollar aproximaciones filogenéticas en cianobacterias (Wilmotte & Herdman, 2001; Svenning, Eriksson, & Rasmussen, 2005; Engene, Cameron & Gerwick, 2010; Bohunická et al., 2015). Estos estudios resaltan la importancia del uso de métodos moleculares asociados a aproximaciones fenotípicas para la correcta identificación taxonómica de las cianobacterias. Además, se propone el uso de genes alternativos de una sola copia como el rpoC1, región espaciadora intergénica de la ficocianina (cpcBA-IGS), genes de la nitrogenasa, entre otros (Seo & Yokota, 2003, Kumari, Srivastava & Bhargava, 2009; Lee et al., 2014). En nuestro estudio, el uso taxonómico de la región de la subunidad gamma de la ARN polimerasa dependiente de ADN (rpoC) no logró esclarecer la separación taxonómica de los cuatros géneros analizados, obteniéndose discrepancias de posicionamiento dentro de la topología construida, ya que las cepas se localizaron dentro un gran grupo del género Nostoc sp., debido a la carencia de secuencias de la región rpoC en el Genbank de los géneros en estudio, además de la posibilidad de amplificación preferencial de ADN contaminante dentro de la muestra (Lee et al., 2014).
Dentro de los géneros identificados, Calothrix sp. se ha utilizado en la estimulación de la producción de amonio y compuestos indólicos (Dhar, Prasanna, Pabbi & Vishwakarma, 2015). Estudios realizados en la determinación de grupos microbianos en campos de leguminosas, se detectaron cepas de dicho género, lo que sugiere que este puede contribuir con la fertilidad de los suelos cultivados de este tipo, como producto de la fijación de nitrógeno (Avendaño, Pérez & Rodríguez, 2014).
Se han reportado especies del género Tolypothrix (T. tenuis) como una especie extensivamente usada como inóculo en los campos tropicales de arroz (Debnath & Bhadury, 2016). Particularmente en Argentina, esta ha sido utilizada en combinación con otros géneros para la producción de biofertilizantes secos adecuados para la aplicación aérea, aportando ventajas tanto al campo de la agricultura como el industrial (Silva & de Jesús, 2013).
Por otra parte, los géneros Nostoc y Tolypothrix, entre otros, se consideran recursos potenciales para la producción de biodiesel como combustible alternativo (Singh et al., 2017). Esto se debe a su capacidad productora de lípidos y a la rápida producción de biomasa, ya que en comparación con otras materias primas consideradas para producción de biodiesel como: aceites vegetales, grasa animal y desechos de aceite de cocina, estas presentan un suministro limitado, lo que impide la expansión de la producción del biocombustible (Prabakaran & Ravindran, 2011).
A partir de cianobacterias del género Scytonema se encontró el pigmento llamado scytonemina, el cual es de valor farmacológico (Bultel-Poncé, Felix-Theodose, Sarthou, Ponge & Bodo, 2004). El cual presenta, propiedades de protección contra la luz ultravioleta, además de anti-inflamatorias y anti-proliferativas en el caso de células T humanas con leucemia. Este pigmento se encuentra en varios géneros de cianobacterias, sin embargo, difieren en cantidad y rendimiento (Singh, Kumari, Rastogi, Sinha & Sinha, 2010).
En cuanto a Nostoc sp., la mayoría de las cianobacterias fotobiontes pertenecen a este género y se caracteriza por ser especies cosmopolitas, bien adaptadas a ambientes terrestres (Arima et al., 2012). En la industria alimenticia, este género juega un papel fisiológico y nutricional muy importante en la dieta humana por su alto contenido de fibra y proteína, y el mismo es consumido en países como Chile, México, Perú y Filipinas (Thajuddin & Subramanian, 2010).
Entre los géneros que han sido utilizados en agricultura reconocidos por su capacidad de mejorar la fertilidad del suelo con la fijación de nitrógeno y biorremediación, el género Nostoc, ha generado resultados prometedores como biofertilizantes útiles en campos experimentales de cultivo de arroz, incluso en zonas afectadas por inundaciones (Avendaño et al., 2014).
Diversos estudios han concluido que las cianobacterias, las cuales poseen potenciales de acondicionamiento de suelos y conservación del ambiente, podrían ser de los candidatos más apropiados en el desarrollo de prácticas sustentables en agricultura, y con ello la minimización de la dependencia de fertilizantes químicos (Kishore & Bimal, 2010).
Para efectos del monitoreo de aplicaciones biotecnológicas evitando bioensayos laboriosos, se ha implementado la identificación directa de genes relacionados a dichos potenciales. Un ejemplo de esto es la amplificación de genes que codifican para enzimas involucradas en procesos metabólicos; por ejemplo, la amplificación de la enzima nitrogenasa a partir de mRNA, la cual se ha realizado en estudios de asimilación de nitrógeno in situ con microorganismos (Mouser et al., 2009; Kumari et al., 2009).
En cuanto al aprovechamiento de cianobacterias como recurso biológico para prácticas en agricultura y otros, existe la necesidad de una rápida identificación y caracterización de taxones capaces de adaptarse a condiciones ambientales. Según nuestros hallazgos, es posible la identificación molecular exitosa de cepas de cianobacterias aisladas de ambientes tropicales mediante una secuencia parcial de la región ribosomal 16S, con el fin de continuar con el estudio de genes responsables de propiedades de interés biotecnológico para así evaluar su potencial hacia futuras aplicaciones y conservación de la biodiversidad.
Agradecimientos
Al Laboratorio de Biotecnología de Microalgas de la Universidad Nacional por brindarnos el material biológico analizado en este estudio. A Narcy Villalobos Sandí y Rebeca Rojas Alfaro por facilitarnos el cultivo y crecimiento de las cepas caracterizadas en este trabajo. A Walter Peraza Padilla de la Escuela de Ciencias Agrarias, UNA, por facilitarlos el equipo de microscopia óptica.
REFERENCIAS
Altschul, S. F., Gish, W., Miller, W., Myers, E. W., & Lipman, D. J. (1990). Basic local alignment search tool. Journal of molecular biology, 215(3), 403-410. doi: 10.1016/S0022-2836(05)80360-2
Arima, H., Horiguchi, N., Takaichi, S., Kofuji, R., Ishida, K. I., Wada, K., & Sakamoto, T. (2012). Molecular genetic and chemotaxonomic characterization of the terrestrial cyanobacterium Nostoc commune and its neighboring species. FEMS microbiology ecology, 79(1), 34-45. doi: 10.1111/j.1574-6941.2011.01195.x
Avendaño, E. M., Pérez, R. M., & Rodríguez, G. S. (2014). Aislamiento, cultivo, viabilidad y evaluación de un consorcio cianobacteria-microalga como acondicionador de suelos. Medicina y Ciencias Biológicas, 35, 51.
Bohunická, M., Pietrasiak, N., Johansen, J. R., Gómez, E. B., Hauer, T., Gaysina, L. A., & Lukešová, A. (2015). Roholtiella, gen. nov. (Nostocales, Cyanobacteria) a tapering and branching cyanobacteria of the family Nostocaceae. Phytotaxa, 197(2), 84-103. doi: 10.11646/phytotaxa.197.2.2
Bultel-Poncé, V., Félix-Théodose, F., Sarthou, C., Ponge, J. F., & Bodo, B. (2004). New pigments from the terrestrial cyanobacterium Scytonema sp. collected on the Mitaraka Inselberg, French Guyana. Journal of natural products, 67(4), 678-681. doi: 10.1021/np034031u
Castresana, J. (2002). Gblocks, v. 0.91b versión en línea. Recuperada de: http://molevol.cmima.csic.es/castresana/Gblocks_server.html
Cuevas, C., Goyenechea, I., & Iturbe U. (2007). La sistemática, base del conocimiento de la biodiversidad. A. Contreras Ramos (Ed.). UAEH.
Darriba, D., Taboada, G. L., Doallo, R., & Posada, D. (2012). jModelTest 2: more models, new heuristics and parallel computing. Nature Methods, 9(8), 772. doi: 10.1038/nmeth.2109
De, P. K. (1939). The role of blue-green algae in nitrogen fixation in rice-fields. Proceedings of the Royal Society of London. Series B, Biological Sciences, 121-139. doi: 10.1098/rspb.1939.0014
Debnath, M., & Bhadury, P. (2016). Adaptive responses and arsenic transformation potential of diazotrophic Cyanobacteria isolated from rice fields of arsenic affected Bengal Delta Plain. Journal of Applied Phycology, 28(5), 2777-2792. doi: 10.1007/s10811-016-0820-9
Desikachary, T. V. (1959). Cyanophyta. Indian Council of Agricultural Research, New Delhi, India. Monographs on Algae, 686.
Dhar, D. W., Prasanna, R., Pabbi, S., & Vishwakarma, R. (2015). Significance of cyanobacteria as inoculants in agriculture. In Algal Biorefinery: An Integrated Approach, 339-374. doi: 10.1007/978-3-319-22813-6_16
Engene, N., Cameron, R., & Gerwick, W. H. (2010). 16S rRNA Gene heterogeneity in the filamentous marine cyanobacterial genus LYNGBYA1. Journal of Phycology, 46(3), 591-601. doi: 10.1111/j.1529-8817.2010.00840.x
Faldu, N., Patel, S., Vishwakarma, N. P., Singh, A. K., Patel, K., & Pandhi, N. (2014). Genetic diversity of marine and fresh water cyanobacteria from the Gujarat state of India. Advances in Bioscience and Biotechnology, 5(14), 1061. doi: 10.4236/abb.2014.514121
Flores, E., López-Lozano, A., & Herrero, A. (2015). Nitrogen Fixation in the Oxygenic Phototrophic Prokaryotes (Cyanobacteria): The Fight Against Oxygen. Biological Nitrogen Fixation, 879-890. doi: 10.1002/9781119053095.ch86
Galhano, V., De Figueiredo, D. R., Alves, A., Correia, A., Pereira, M. J., Gomes-Laranjo, J., & Peixoto, F. (2011). Morphological, biochemical and molecular characterization of Anabaena, Aphanizomenon and Nostoc strains (Cyanobacteria, Nostocales) isolated from Portuguese freshwater habitats. Hydrobiología, 663(1), 187-203. Doi: 10.1007/s10750-010-0572-5
Gugger, M., Lyra, C., Suominen, I., Tsitko, I., Humbert, J. F., Salkinoja-Salonen, M. S., & Sivonen, K. (2002). Cellular fatty acids as chemotaxonomic markers of the genera Anabaena, Aphanizomenon, Microcystis, Nostoc and Planktothrix (cyanobacteria). International Journal of Systematic and Evolutionary Microbiology, 52(3), 1007-1015. doi: 10.1099/ijs.0.01917-0
Gupta, M., & Chaudhuri, A. N. (2016). Bioremediation of cadmium and lead and its effect on chlorophyll, enzymatic and non enzymatic antioxidants of cyanobacteria. International Journal of Scientific Research, 4(6), 6-10.
Iteman, I., Rippka, R., de Marsac, N. T., & Herdman, M. (2002). rDNA analyses of planktonic heterocystous cyanobacteria, including members of the genera Anabaenopsis and Cyanospira. Microbiology, 148(2), 481-496. doi: 10.1099/00221287-148-2-481
Jensen, S., Petersen, B. O., Omarsdottir, S., Paulsen, B. S., Duus, J. Ø., & Olafsdottir, E. S. (2013). Structural characterisation of a complex heteroglycan from the cyanobacterium Nostoc commune. Carbohydrate polymers, 91(1), 370-376. doi: 10.1016/j.carbpol.2012.08.063
Kishore, K., & Bimal, R. (2010). Distribution of nitrogen-fixing cyanobacteria (Nostocaceae) during rice cultivation in fertilized and unfertilized paddy fields. Nordic Journal of Botany, 28(1), 100-103. doi: 10.1111/j.1756-1051.2009.00486.x
Komárek, J., & Anagnostidis, K. (1989). Modern approach to the classification system of cyanophytes-4: Nostocales. Archives of Hydrobiology, Suppl. Algological Studies, 56, 247-345.
Komárek, J., & Anagnostidis, K. (1999). Cyanoprokaryota: I. Teil Chroococcales. Band 19/1, Sußwasserflora von Mitteleuropa, Jena: Gaustav Fischer.
Komárek, J., & Anagnostidis, K. (2005). Cyanoprokaryota II. Teil Oscillatoriales. Band 19/2, Süßwasserflora von Mitteleuropa, Munchen: Elsevier GmbH,.
Komarek, J., Sant’Anna, C. L., Bohunická, M., Mareš, J., Hentschke, G. S., Rigonato, J., & Fiore, M. F. (2014). Phenotype diversity and phylogeny of selected Scytonema-species (Cyanoprokaryota) from SE Brazil. Fottea, 13(2), 173-200.
Kumari, N., Srivastava, A. K., & Bhargava, P. (2009). Molecular approaches towards assessment of cyanobacterial biodiversity. African Journal of Biotechnology, 8(18), 4284-4298.
Lee, E., Ryan, U. M., Monis, P., McGregor, G. B., Bath, A., Gordon, C., & Paparini, A. (2014). Polyphasic identification of cyanobacterial isolates from Australia. Water Research, 59, 248-261. doi: 10.1016/j.watres.2014.04.023
Liao, H. F., Wu, T. J., Tai, J. L., Chi, M. C., & Lin, L. L. (2015). Immunomodulatory potential of the polysaccharide-rich extract from edible cyanobacterium Nostoc commune. Medical Sciences, 3(4), 112-123. doi: 10.3390/medsci3040112
Lyra, C., Suomalainen, S., Gugger, M., Vezie, C., Sundman, P., Paulin, L., & Sivonen, K. (2001). Molecular characterization of planktic cyanobacteria of Anabaena, Aphanizomenon, Microcystis and Planktothrix genera. International Journal of Systematic and Evolutionary Microbiology, 51(2), 513-526. doi: 10.1099/00207713-51-2-513
Mouser, P. J., N’Guessan, A. L., Elifantz, H., Holmes, D. E., Williams, K. H., Wilkins, M. J. & Lovley, D. R. (2009). Influence of heterogeneous ammonium availability on bacterial community structure and the expression of nitrogen fixation and ammonium transporter genes during in situ bioremediation of uranium-contaminated groundwater. Environmental Science & Technology, 43(12), 4386-4392. doi: 10.1021/es8031055
Porebski, S., Bailey, L. G., & Baum, B. R. (1997). Modification of a CTAB DNA extraction protocol for plants containing high polysaccharide and polyphenol components. Plant Molecular Biology Reporter, 15(1), 8-15. doi: 10.1007/BF02772108
Prabakaran, P., & Ravindran, A. D. (2011). A comparative study on effective cell disruption methods for lipid extraction from microalgae. Letters in Applied Microbiology, 53(2), 150-154. doi: 10.1111/j.1472-765X.2011.03082.x
Prasanna, R., Sood, A., Ratha, S. K., & K Singh, P. (2013). Cyanobacteria as a “green” option for sustainable agriculture. Cyanobacteria: An Economic Perspective, 145-166. doi: 10.1002/9781118402238.ch9
Quesada, A., & Vincent, W. F. (2012). Cyanobacteria in the cryosphere: snow, ice and extreme cold. In Ecology of Cyanobacteria II. pp. 387-399. doi: 10.1007/978-94-007-3855-3_14
Rajaniemi, P., Hrouzek, P., Kaštovska, K., Willame, R., Rantala, A., Hoffmann, L. & Sivonen, K. (2005). Phylogenetic and morphological evaluation of the genera Anabaena, Aphanizomenon, Trichormus and Nostoc (Nostocales, Cyanobacteria). International Journal of Systematic and Evolutionary Microbiology, 55(1), 11-26. doi: 10.1099/ijs.0.63276-0
Rambaut, A. (2009). FigTree v1.4 2012–2014: Tree Figure Drawing Tool. Recuperado de: http://tree.bio.ed.ac.uk/software/figtree.
Rinkel, B. E., & Manoylov, K. M. (2014). Calothrix an evaluation of fresh water species in United States rivers and streams, their distribution and preliminary ecological findings. Proceedings of the academy of natural sciences of Philadelphia, 163(1), 43-59. doi: 10.1635/053.163.0108
Sant’Anna, C. L., Azevedo, M. T. P., Fiore, M. F., Lorenzi, A. S., Kaštovský, J., & Komárek, J. (2011). Subgeneric diversity of Brasilonema (Cyanobacteria, Scytonemataceae). Brazilian Journal of Botany, 34(1), 51-62.
Seo, P. S., & Yokota, A. (2003). The phylogenetic relationships of cyanobacteria inferred from 16S rRNA, gyrB, rpoC1 and rpoD1 gene sequences. The Journal of general and applied microbiology, 49(3), 191-203. doi: 10.2323/jgam.49.191
Sharma, N. K. (2015). From natural to human-impacted ecosystems: rationale to investigate the impact of urbanization on cyanobacterial diversity in soils. Biodiversity and Conservation, 24(4), 1007-1015. doi: 10.1007/s10531-015-0897-5
Sihvonen, L. M., Lyra, C., Fewer, D. P., Rajaniemi-Wacklin, P., Lehtimäki, J. M., Wahlsten, M., & Sivonen, K. (2007). Strains of the cyanobacterial genera Calothrix and Rivularia isolated from the Baltic Sea display cryptic diversity and are distantly related to Gloeotrichia and Tolypothrix. FEMS microbiology ecology, 61(1), 74-84. doi: 10.1111/j.1574-6941.2007.00321.x
Silva, P. G., & de Jesús S. H. (2013). Biomass production of Tolypothrix tenuis as a basic component of a cyanobacterial biofertilizer. Journal of Applied Phycology, 25(6), 1729-1736. doi: 10.1007/s10811-013-0035-2
Singh, S. P., Kumari, S., Rastogi, R. P., Sinha, R., & Sinha, R. P. (2010). Photoprotective and biotechnological potentials of cyanobacterial sheath pigment, scytonemin. African Journal of Biotechnology, 9(5), 580-588.
Singh, S. P., Pathak, J., & Sinha, R. P. (2017). Cyanobacterial factories for the production of green energy and value-added products: An integrated approach for economic viability. Renewable and Sustainable Energy Reviews, 69, 578-595. doi: 10.1016/j.rser.2016.11.110
Stamatakis, A., Ludwig, T., & Meier, H. (2005). RAxML-III: a fast program for maximum likelihood-based inference of large phylogenetic trees. Bioinformatics, 21(4), 456-463. doi: 10.1093/bioinformatics/bti191
Svenning, M. M., Eriksson, T., & Rasmussen, U. (2005). Phylogeny of symbiotic cyanobacteria within the genus Nostoc based on 16S rDNA sequence analyses. Archives of microbiology, 183(1), 19-26 doi. 10.1007/s00203-004-0740-y
Teneva, I., Stoyanov, P., Mladenov, R., & Dzhambazov, B. (2012). Molecular and phylogenetic characterization of two species of the genus Nostoc (Cyanobacteria) based on the cpcB-IGS-cpcA locus of the phycocyanin operon. Journal of Bioscience and Biotechnology, 1(1), 9-19.
Thajuddin, N., & Subramanian, G. (2010). Cyanobacterial biodiversity and potential applications in biotechnology. Current Science, 89(1), 47-57.
Wilmotte, A., and M. Herdman. 2001. Phylogenetic relationships among cyanobacteria based on 16S rRNA sequences, pp. 487-493. In D. R. Boone and R. W. Castenholz (ed.), Bergey’s Manual of Systematic Bacteriology, vol. 1. New York, N.Y.: Springer.
Zhen, Z., Liu, J., Rensing, C., Yan, C., & Zhang, Y. (2017). Effects of two different high-fidelity DNA polymerases on genetic analysis of the cyanobacterial community structure in a subtropical deep freshwater reservoir. Archives of Microbiology, 199(1), 125-134. doi: 10.1007/s00203-016-1284-7
Fig. 1. Cepas de cianobacterias aisladas de diferentes ecosistemas tropicales. (A) Calothrix sp. (BGN17), (B) Calothrix sp. (BGN18) (C) Scytonema sp. (BGN23), (D) Tolypothrix sp. (BGN21), (E) Nostoc sp. (BGN04), (F) Nostoc sp. (BGN25).

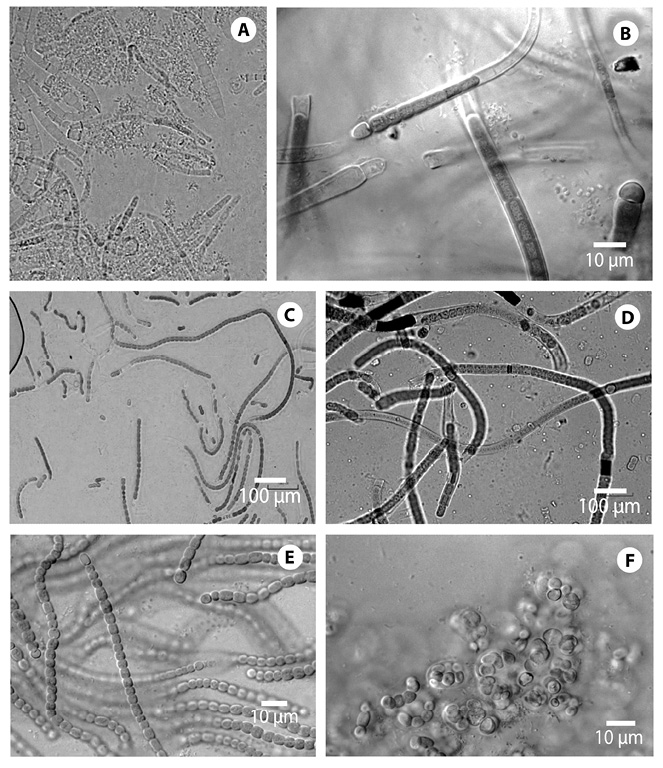
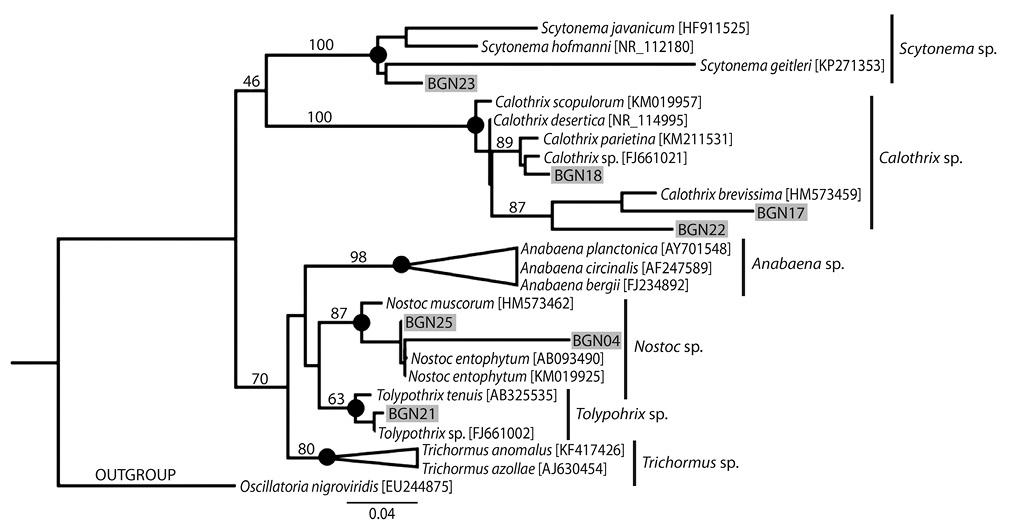
Fig. 2. Árbol de posicionamiento filogenético de cianobacterias tropicales, mediante máxima verosimilitud (ML) y basado en secuencias parciales de la subunidad pequeña del ARN ribosomal (16S ARNr). Las cepas de este estudio (BGN#) están marcadas con un cuadrado gris. El número en las ramas indica el valor de soporte boostrap (porcentaje derivado de 2 000 repeticiones). Valores menores al 45% no se muestran. Oscillatoria nigroviridis es definida como grupo externo (outgroup). Los números de accesión se muestran entre paréntesis para las secuencias obtenidas del Genbank.